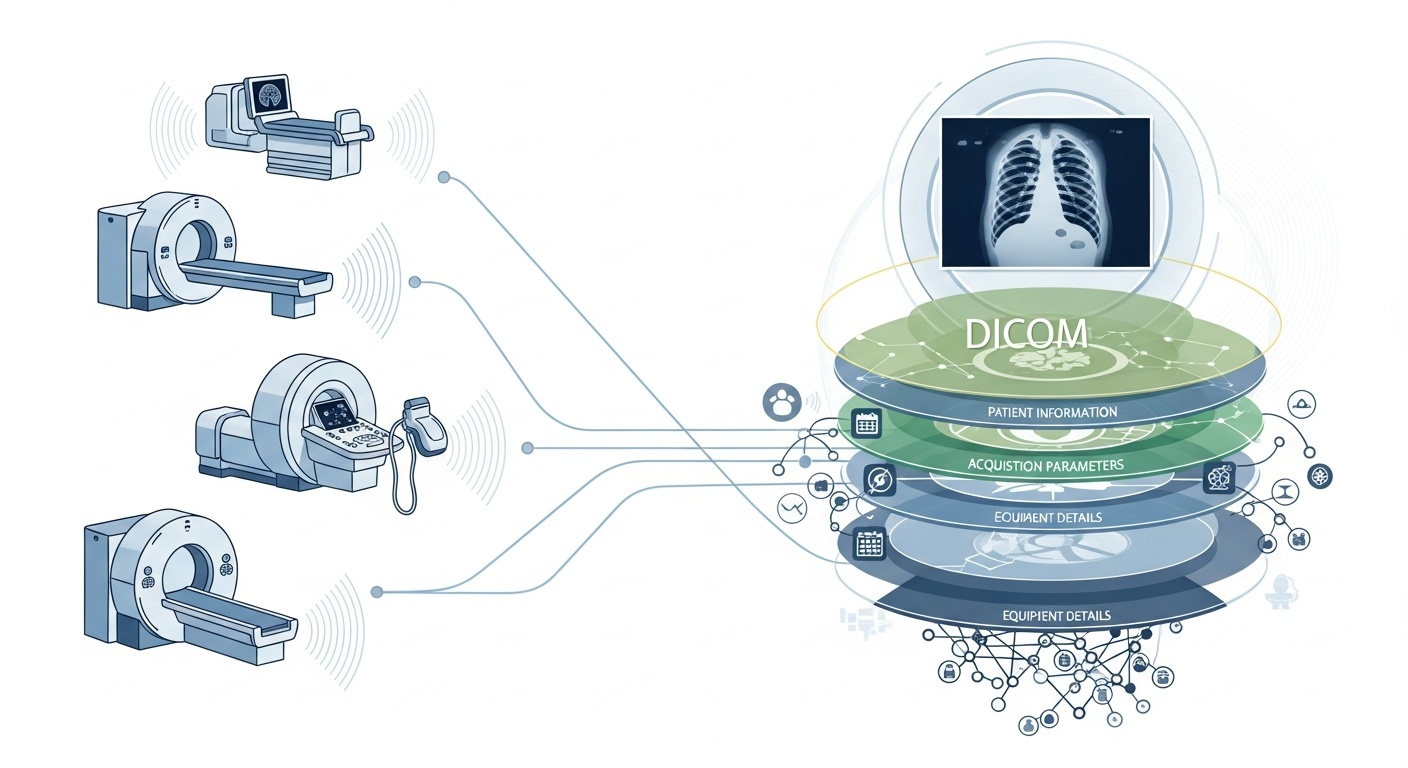
Cada vez que un equipo de radiología escanea a un paciente, por ejemplo, una tomografía computarizada, una resonancia magnética o una ecografía, se genera una cascada de datos. Lo más visible es la imagen en sí, pero detrás de ella hay una rica capa de metadatos: el quién, el qué, el cuándo, el cómo y el dónde de la tomografía. Esa capa se rige por el estándar DICOM (Digital Imaging and Communications in Medicine), un formato de imágenes médicas que la Asociación Nacional de Fabricantes de Productos Eléctricos (NEMA) y el Colegio Estadounidense de Radiología (ACR) crearon hace décadas.
Lo que hace que los metadatos sean tan interesantes es que están estructurados, son legibles por máquinas y son extraordinariamente detallados: la configuración del equipo, los parámetros de adquisición, la demografía de los pacientes, las identificaciones de los estudios e incluso los códigos de las instituciones y los detalles del fabricante de la modalidad. Esa riqueza es la que permite el análisis de macrodatos, la investigación posterior, la modelización de la IA y la estandarización de los protocolos, si se aprovecha bien.
En el contexto de los macrodatos, no estamos hablando solo de unas pocas docenas de estudios de imágenes. Estamos hablando de cientos de miles, o incluso millones, de imágenes de diferentes modalidades, sitios y proveedores, con los metadatos como la capa clave de indexación: el «quién, cuándo y cómo» de cada elemento. Si no se utilizan los metadatos de forma eficaz, se corre el riesgo de tener un archivo enorme de imágenes, pero con una capacidad mínima para consultarlas, compararlas o obtener información a partir de ellas. En una revisión reciente se afirma que «nunca se vuelve a acceder a la mayoría de la información almacenada en los archivos del PACS», una oportunidad desperdiciada.
Supongamos que está realizando un estudio en varios sitios de tomografías computarizadas pulmonares para la detección temprana del enfisema. Deberás seleccionar las tomografías en función de los parámetros: el fabricante, la marca o el modelo del escáner, el grosor de la rebanada, el núcleo de la reconstrucción, la edad del paciente, los intervalos de fechas e incluso los parámetros de dosis. La mayoría de ellos son campos de metadatos, no datos de píxeles. La extracción de esas etiquetas permite crear la cohorte, excluir los escaneos incompatibles (por ejemplo, cortes demasiado gruesos) y garantizar la comparabilidad.
Los metadatos permiten supervisar el proceso de obtención de imágenes en sí: ¿la institución utiliza el protocolo correcto? ¿Los parámetros de adquisición se desvían con el tiempo (por ejemplo, el campo de visión, el tiempo de inyección de contraste)? ¿La configuración del proveedor es coherente? En un mundo con grandes volúmenes de datos en el que se realizan miles de escaneos al día, no se puede confiar en la observación ocular humana. Es necesario analizar los metadatos. Por este motivo, muchos sistemas PACS no explotan suficientemente los metadatos.
Si construyes una canalización de IA o radiómica, no puedes tratar todas las imágenes como intercambiables. Los metadatos se convierten en una variable de control integral: las funciones de entrada suelen incluir la modalidad, el kVp (pico de kilovoltaje), el fabricante, el núcleo, el grosor del corte e incluso la fecha o el hospital (cambio de dominio). Estos campos de metadatos ayudan a gestionar los sesgos, armonizar los datos y anotar las imágenes según el contexto. Muchos investigadores consideran que los metadatos son «tan importantes como los datos en píxeles».
Big data significa escalabilidad. Esto implica fuentes variadas, múltiples proveedores, diferentes instituciones y formatos heterogéneos. Los metadatos DICOM son el «lenguaje» estandarizado que ayuda a unificar la capa de metadatos, permitir la búsqueda y la indexación, garantizar la interoperabilidad y crear arquitecturas escalables (PACS en la nube, archivos federados). Sin embargo, la implementación es importante. El mismo estudio de PMC descubrió que muchos sistemas no aprovechan al máximo el estándar.
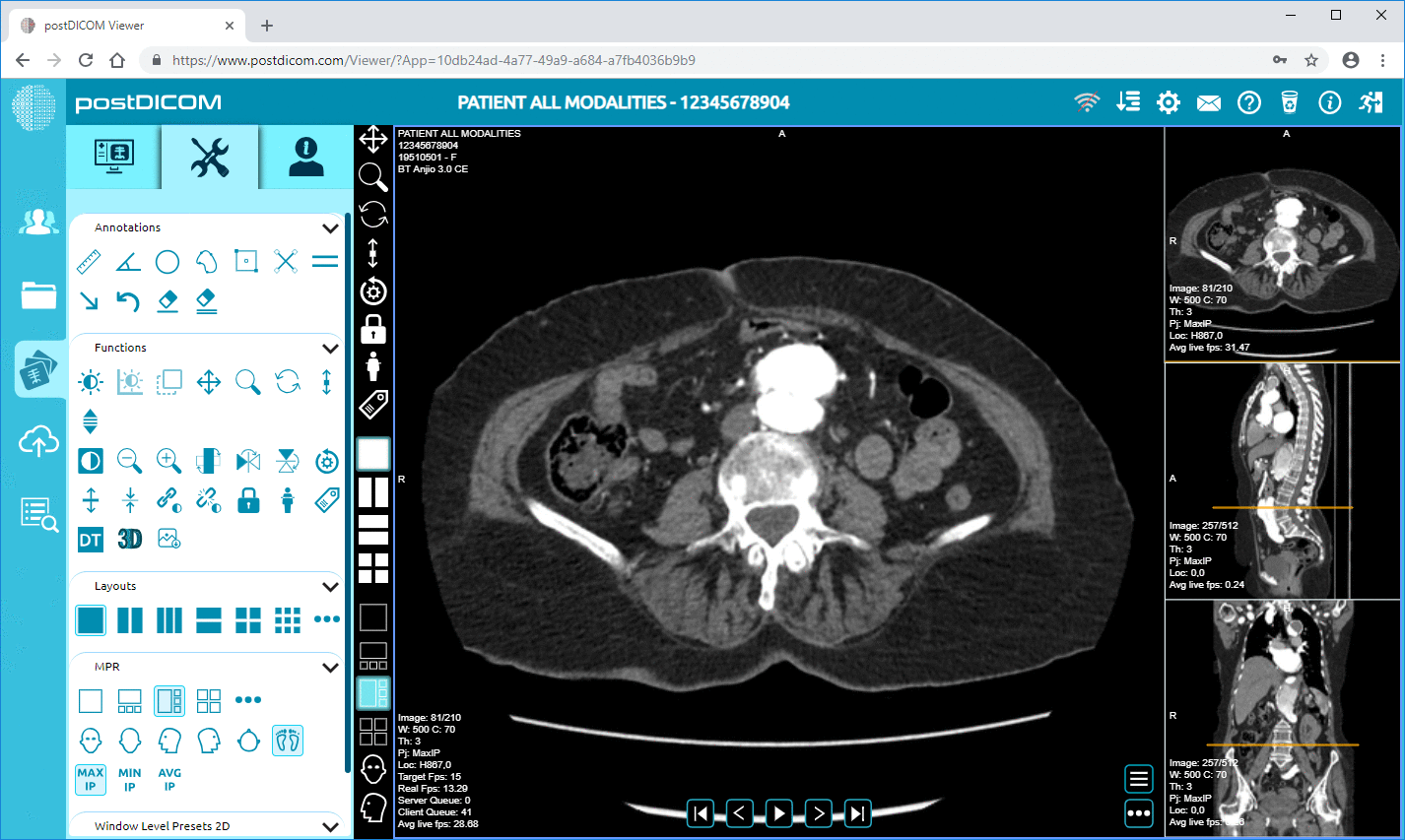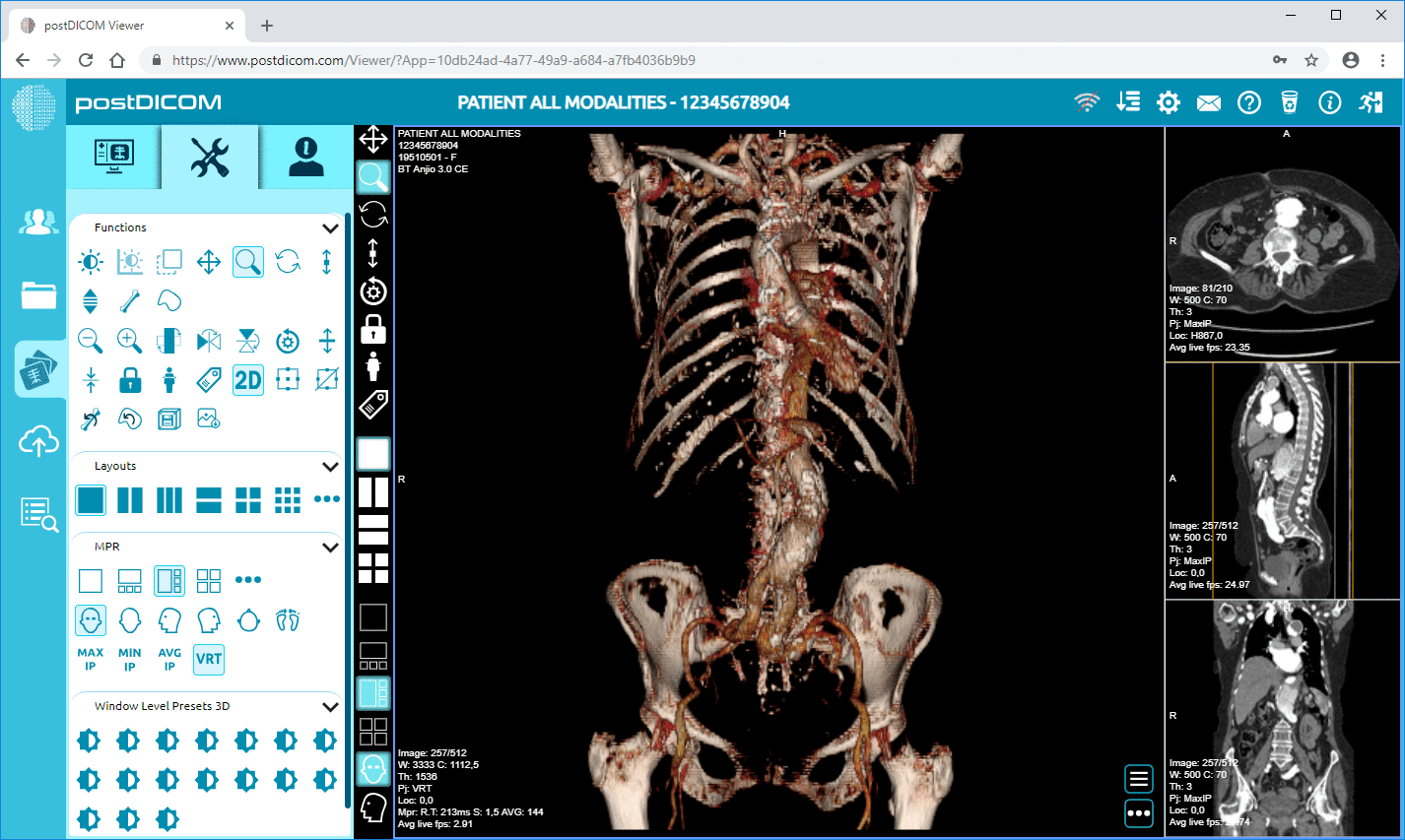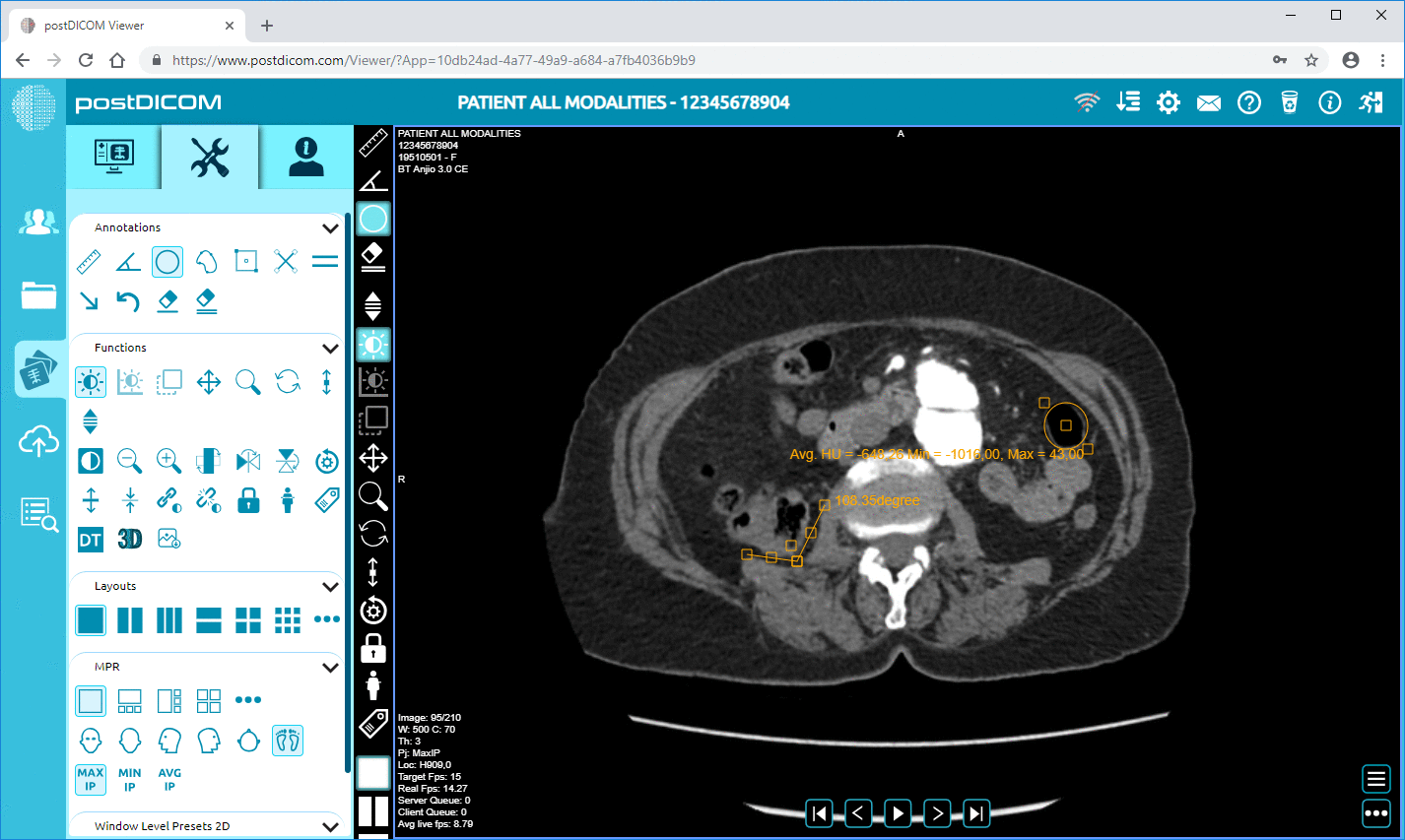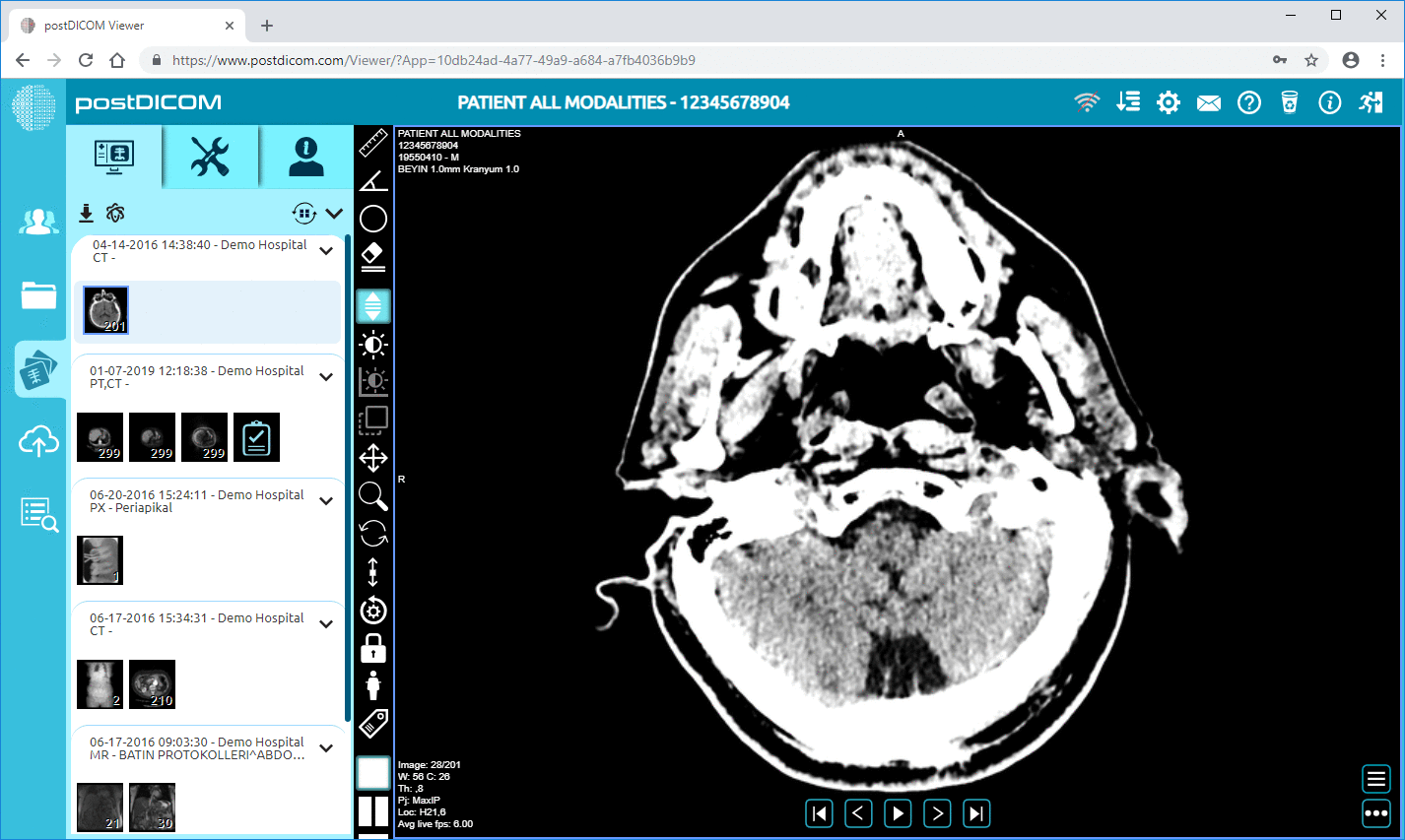
PostDicom ofrece un PACS (sistema de comunicación y archivo de imágenes) basado en la nube para almacenar, ver y compartir estudios de imágenes y documentos clínicos. Algunas características clave de PostDicom relacionadas con los metadatos:
• Soporte para etiquetas y descripciones DICOM: nuestra biblioteca de recursos incluye «Modalidad y etiquetas DICOM», lo que permite a los usuarios acceder a listas de etiquetas y descripciones. Modalidad y etiquetas DICOM
• Integración entre API y Fhir: admite las interfaces API y FHIR (Fast Healthcare Interoperability Resources), lo que permite consultar los metadatos mediante programación, integrarlos con otros sistemas y analizarlos.
• Escalabilidad en la nube y uso compartido en varios sitios: compartir entre pacientes, médicos e instituciones; la escalabilidad ilimitada significa que los canales de big data son factibles.
• Procesamiento avanzado de imágenes y compatibilidad multimodal: si bien esto se refiere a los píxeles, la compatibilidad con modalidades como la PET—CT y las series múltiples significa que los metadatos son sustanciales (valores de SUV, volúmenes de fusión, tipo de modalidad) y están disponibles para el análisis.
El uso de una plataforma como PostDicom le permite aprovechar los metadatos a través de flujos de trabajo estructurados, API y arquitectura en la nube.
A continuación, se explica cómo estructurar el flujo de trabajo, desde archivos sin procesar hasta información lista para el análisis.
 - Created by PostDICOM.jpg)
El primer paso para aprovechar los metadatos DICOM para el análisis es la extracción y la normalización. Las bibliotecas, como el paquete PyDICOM de código abierto para Python, se suelen utilizar para analizar archivos DICOM y extraer las etiquetas pertinentes, incluidas las filas y columnas de imágenes, los núcleos de convolución y los parámetros de adquisición específicos de cada modalidad.
Manejar la heterogeneidad es crucial, ya que los diferentes proveedores suelen utilizar etiquetas privadas o implementaciones no estándar. Para garantizar la coherencia entre los conjuntos de datos, se requieren un análisis sólido, una lógica alternativa y tablas completas de mapeo de etiquetas.
Una vez extraídos, los metadatos deben normalizarse y asignarse a ontologías y estructuras estándar, como códigos de modalidad, nombres de proveedores, categorías de grosor de corte y formatos de fecha y hora estandarizados.
Por último, los metadatos estructurados deben almacenarse en un entorno de big data, como una base de datos relacional, un almacén NoSQL o un lago de datos en columnas, con indexación para permitir consultas rápidas y eficientes.
Una vez extraídos, los metadatos deben someterse a un control de calidad para garantizar su precisión y confiabilidad. Los campos que faltan o son inconsistentes, como los valores de grosor de los cortes en blanco, las etiquetas de modalidad inconsistentes o los UID de las instancias de estudio duplicados, deben identificarse y corregirse.
La privacidad y la anonimización también son fundamentales en esta etapa, ya que los metadatos suelen contener información de identificación personal, incluidos los nombres, las identificaciones y las fechas de los pacientes; las herramientas y los protocolos de desidentificación son esenciales.
Mantener registros de auditoría exhaustivos es otra práctica importante, ya que documenta cuándo se extrajeron los metadatos, qué versiones del analizador se usaron y cualquier error encontrado durante el proceso.
Las políticas de gobierno también deben definir los campos obligatorios y proporcionar orientación sobre cómo gestionar los conjuntos de datos heredados o incompletos para garantizar que los análisis posteriores sean precisos y cumplan con las normas.
El siguiente paso es la indexación basada en metadatos y la ingeniería de funciones, que transforma los metadatos sin procesar en información procesable.
Esto implica crear índices y filtros que permitan a los investigadores y analistas consultar conjuntos de datos específicos, por ejemplo, recuperar todas las tomografías computarizadas de tórax con un grosor de corte inferior a 1,5 milímetros de un proveedor en particular dentro de un intervalo de fechas determinado.
La ingeniería de funciones se basa en esto al combinar campos de metadatos como el proveedor, el modelo, la fecha de adquisición, el grosor del corte, el núcleo de convolución, el protocolo de contraste, la región corporal, la dosis de radiación y el ID de la institución en variables estructuradas adecuadas para el análisis.
Los metadatos también se pueden vincular a conjuntos de datos clínicos, conectando los datos de imágenes con los resultados, diagnósticos o tratamientos de los pacientes. Esta vinculación permite una visión más holística de los datos de imágenes y su contexto clínico.
Una vez que se indexan los metadatos y se diseñan las funciones, el análisis y la generación de información son posibles.
El análisis descriptivo puede revelar los volúmenes de estudio por modalidad, proveedor o región, rastrear las tendencias en los parámetros de adquisición y resaltar los errores o inconsistencias en las prácticas de obtención de imágenes. Los análisis comparativos permiten evaluar los protocolos de adquisición en todas las instituciones, detectar desviaciones e identificar los análisis de valores atípicos que pueden requerir una atención especial.
Para las aplicaciones de aprendizaje automático e inteligencia artificial, los metadatos son esenciales para controlar el cambio de dominio, garantizar que los conjuntos de datos de entrenamiento y prueba se estratifiquen adecuadamente y combinar funciones basadas en píxeles con variables de metadatos estructurados. De este modo, los paneles operativos pueden aprovechar estos datos para supervisar la carga de trabajo, evaluar las métricas de garantía de calidad y garantizar el cumplimiento de los protocolos en todos los sitios.
Por último, los comentarios y la mejora continua completan el ciclo de vida de los metadatos. La información obtenida de los análisis puede servir de base para perfeccionar los protocolos de adquisición y estandarizar los flujos de trabajo a fin de mejorar la calidad general de los datos.
Los nuevos estudios de imágenes y metadatos deben ingerirse de forma continua, supervisando el rendimiento del almacén de metadatos, los tiempos de consulta y la integridad de los datos. Las lecciones aprendidas deben archivarse para capturar los campos de metadatos predictivos, abordar las brechas o errores recurrentes y mejorar las prácticas de gobierno.
Este enfoque iterativo garantiza que las canalizaciones de metadatos sigan siendo sólidas, escalables y valiosas para la investigación futura, las aplicaciones de inteligencia artificial y la toma de decisiones operativas.
• Variabilidad entre proveedores e instituciones: etiquetas privadas o interpretaciones estándar poco estrictas.
• Metadatos faltantes o dañados: los estudios más antiguos pueden tener encabezados incompletos.
• Privacidad y anonimización de los datos: la PHI debe estar anonimizada para la investigación en varios sitios.
• Escalabilidad y rendimiento: millones de imágenes requieren un procesamiento y almacenamiento eficientes.
• Cambio de dominio o sesgo: los proveedores y protocolos dominantes pueden sesgar los modelos de IA.
• Cuestiones reglamentarias y de cumplimiento: las implementaciones multirregionales pueden implicar la HIPAA, el RGPD o las normativas locales.
Los metadatos DICOM son el esqueleto oculto del análisis de imágenes. Plataformas como PostDICOM ilustran cómo transformar un archivo fragmentado de archivos DICOM en un ecosistema con capacidad de búsqueda, escalable y basado en metadatos. Si quieres explorar PostDicom, te animamos a que obtengas nuestra prueba gratuita de 7 días.
|
Cloud PACS y visor DICOM en líneaCargue imágenes DICOM y documentos clínicos a los servidores PostDICOM. Almacene, visualice, colabore y comparta sus archivos de imágenes médicas. |