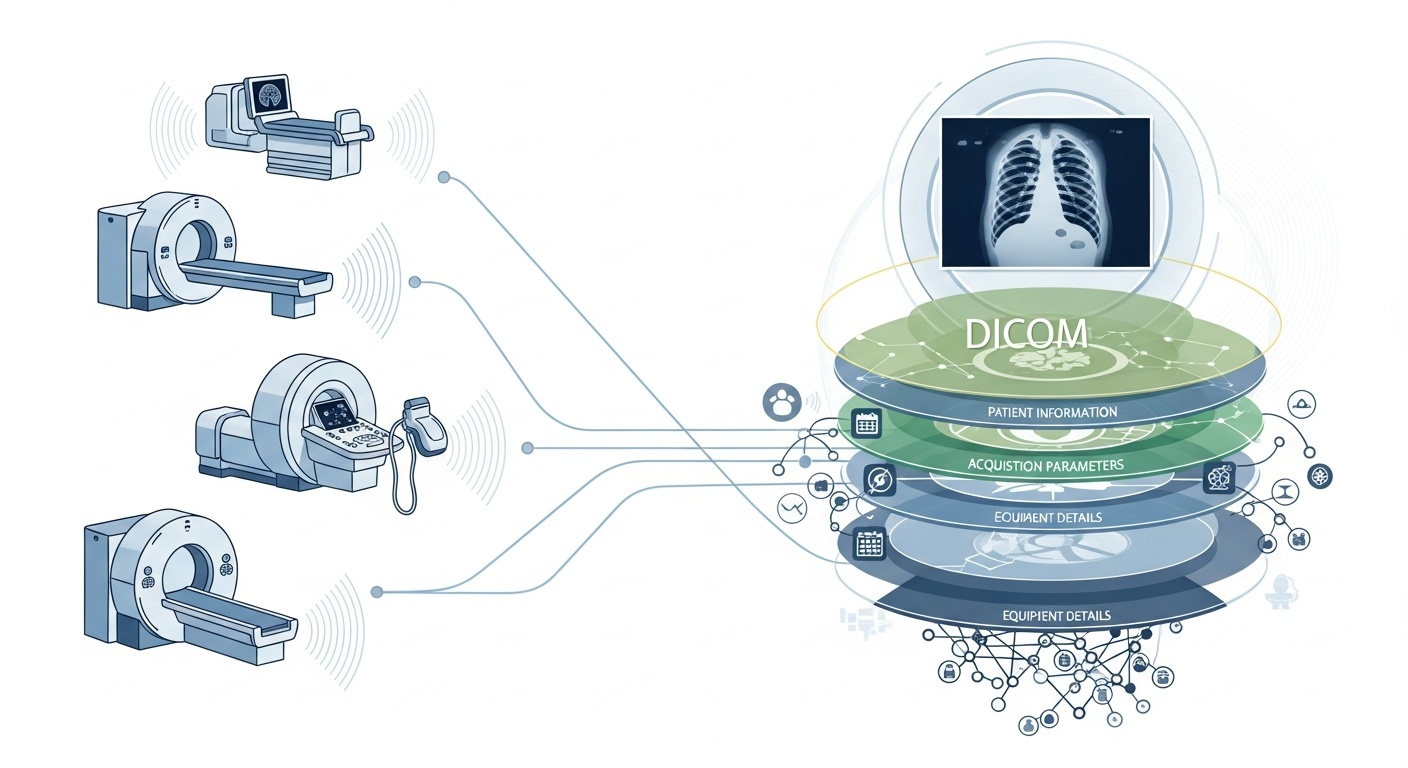
Hver gang et radiologiteam scanner en patient, for eksempel en CT, MR eller ultralyd, genereres en kaskade af data. Mest synligt er selve billedet, men bag det er et rigt lag af metadata: hvem, hvad, hvornår, hvordan og hvor af scanningen. Dette lag styres af standarden DICOM (Digital Imaging and Communications in Medicine), en medicinsk billedformatstandard indbygget af National Electrical Manufacturers Association (NEMA) og American College of Radiology (ACR) for årtier siden.
Hvad der gør metadataene så interessante er, at de er strukturerede, maskinlæsbare og bemærkelsesværdigt detaljerede: udstyrsindstillinger, anskaffelsesparametre, patientdemografi, studie-id'er, endda institutionskoder og modalitetsproducentdetaljer. Denne rigdom er det, der muliggør analyse af store data, downstream-forskning, AI-modellering og protokolstandardisering, hvis du udnytter det godt.
I forbindelse med big data taler vi ikke kun om et par dusin billeddannelsesundersøgelser. Vi taler om hundredtusinder eller endda millioner af billeder på tværs af modaliteter, websteder og leverandører, med metadata som nøgleindekseringslaget: „hvem og hvornår og hvordan“ for hvert element. Uden effektivt at bruge metadata risikerer du at have et massivt arkiv med billeder, men minimal evne til at forespørge, sammenligne eller hente indsigt fra dem. En nylig gennemgang siger: „størstedelen af de oplysninger, der er gemt i PACS-arkiver, er aldrig tilgængelig igen,“ en spildt mulighed.
Sig, at du kører en undersøgelse på flere steder af lunge-CT-scanninger til tidlig detektion af emfysem. Du vil gerne vælge scanninger baseret på parametre: scannerleverandør/mærke/model, skivetykkelse, rekonstruktionskerne, patientens alder, datointervaller, måske endda dosisparametre. De fleste af disse er metadatafelter, ikke pixeldata. Ved at udtrække disse tags kan du opbygge kohorten, udelukke inkompatible scanninger (f.eks. for tykke skiver) og sikre sammenlignelighed.
Metadata giver dig mulighed for at overvåge selve billeddannelsesprocessen: bruger institutionen den korrekte protokol? Afviger anskaffelsesparametre over tid (f.eks. Synsfelt, kontrastinjektionstid)? Er leverandørindstillingerne konsistente? I en verden med store data, hvor tusindvis af scanninger sker om dagen, kan du ikke stole på menneskelig opmærksomhed. Du har brug for analyse af metadata. Mange PACS-systemer underudnytter metadata af denne grund.
Hvis du bygger en AI- eller radiomics-pipeline, kan du ikke behandle hvert billede som udskifteligt. Metadata bliver en integreret kontrolvariabel: inputfunktioner inkluderer ofte modalitet, kVP (kilovoltage-top), producent, kerne, skivetykkelse og endda datoen eller hospitalet kan have betydning (domæneskift). Disse metadatafelter hjælper med at administrere bias, harmonisere data og kommentere billeder med kontekst. Mange forskere kalder metadata „lige så vigtige som pixeldata.“
Big data betyder skala. Det indebærer forskellige kilder, flere leverandører, forskellige institutioner og heterogene formater. DICOM-metadata er det standardiserede „sprog“, der hjælper med at forene metadatalaget, muliggøre søg/indeks, sikre interoperabilitet og opbygge skalerbare arkitekturer (cloud-PAC'er, fødererede arkiver). Men implementering betyder noget. Den samme PMC-undersøgelse viste, at mange systemer ikke udnytter standarden fuldt ud.
PostDiCom tilbyder et skybaseret PACS (Picture Archiving and Communication System) til lagring, visning og deling af billedstudier og kliniske dokumenter. Nogle vigtige metadata-relevante funktioner i PostDiCom:
• Support til Dicom-tags og beskrivelser: Vores ressourcebibliotek viser „DICOM Modality & Tags“, så brugerne kan få adgang til taglister og beskrivelser. DICOM-modalitet og tags
• Api/Fhir-integration: Det understøtter API- og FHIR (Fast Healthcare Interoperability Resources) grænseflader, så metadata kan programmeres, integreres med andre systemer og analyseres.
• Skyskalerbarhed og deling på flere websteder: Deling på tværs af patienter, læger, institutioner; ubegrænset skalerbarhed betyder, at store datapipeliner bliver mulige.
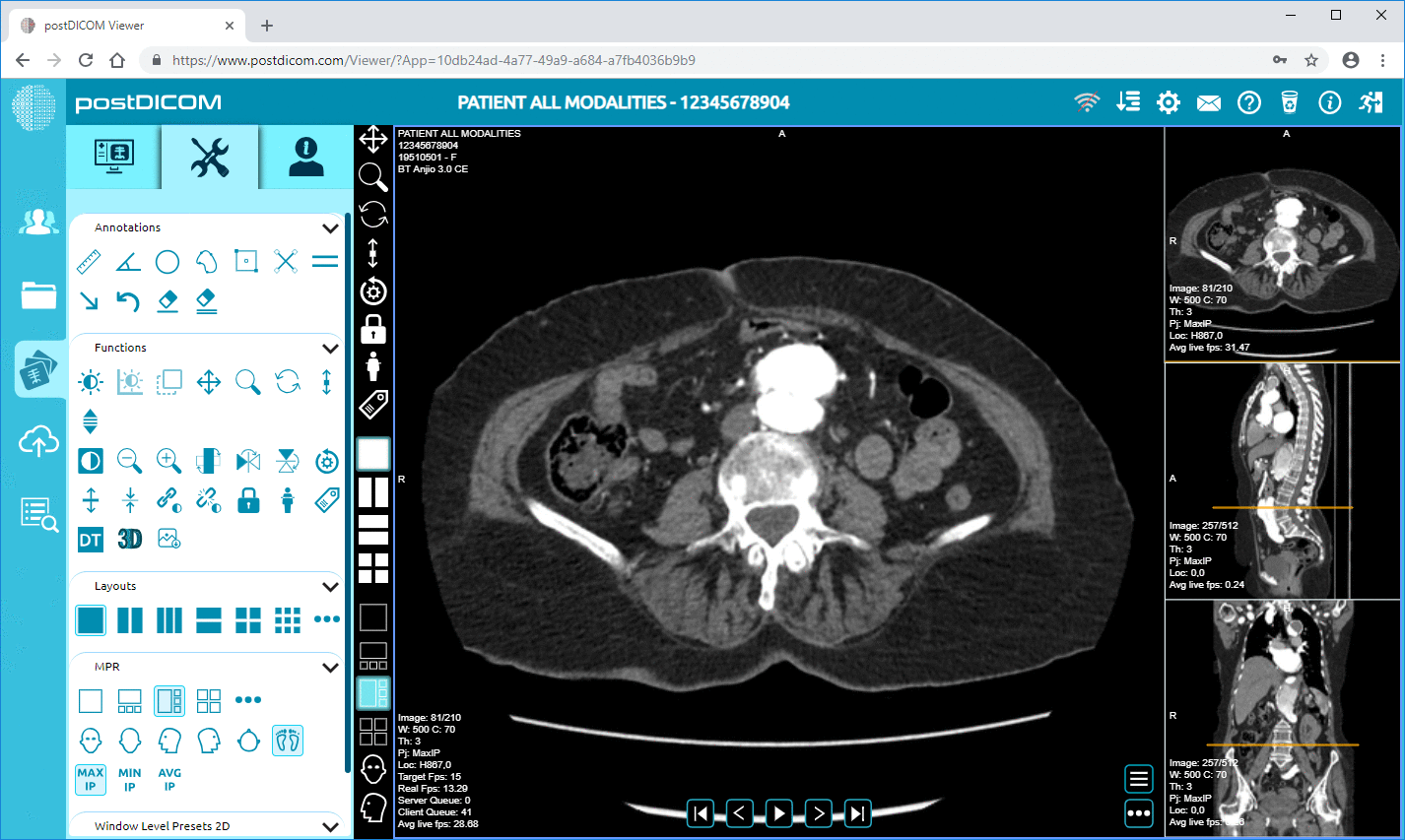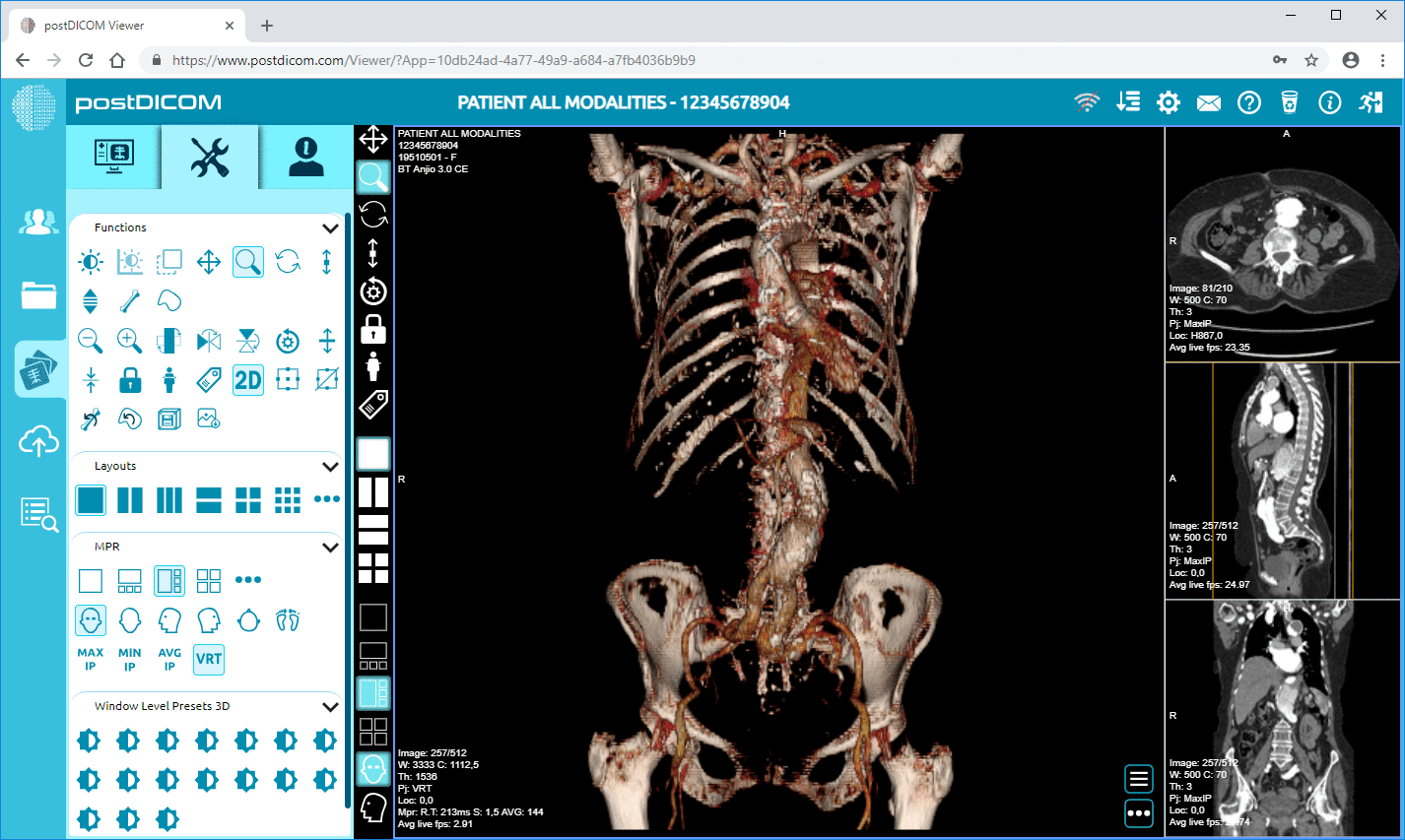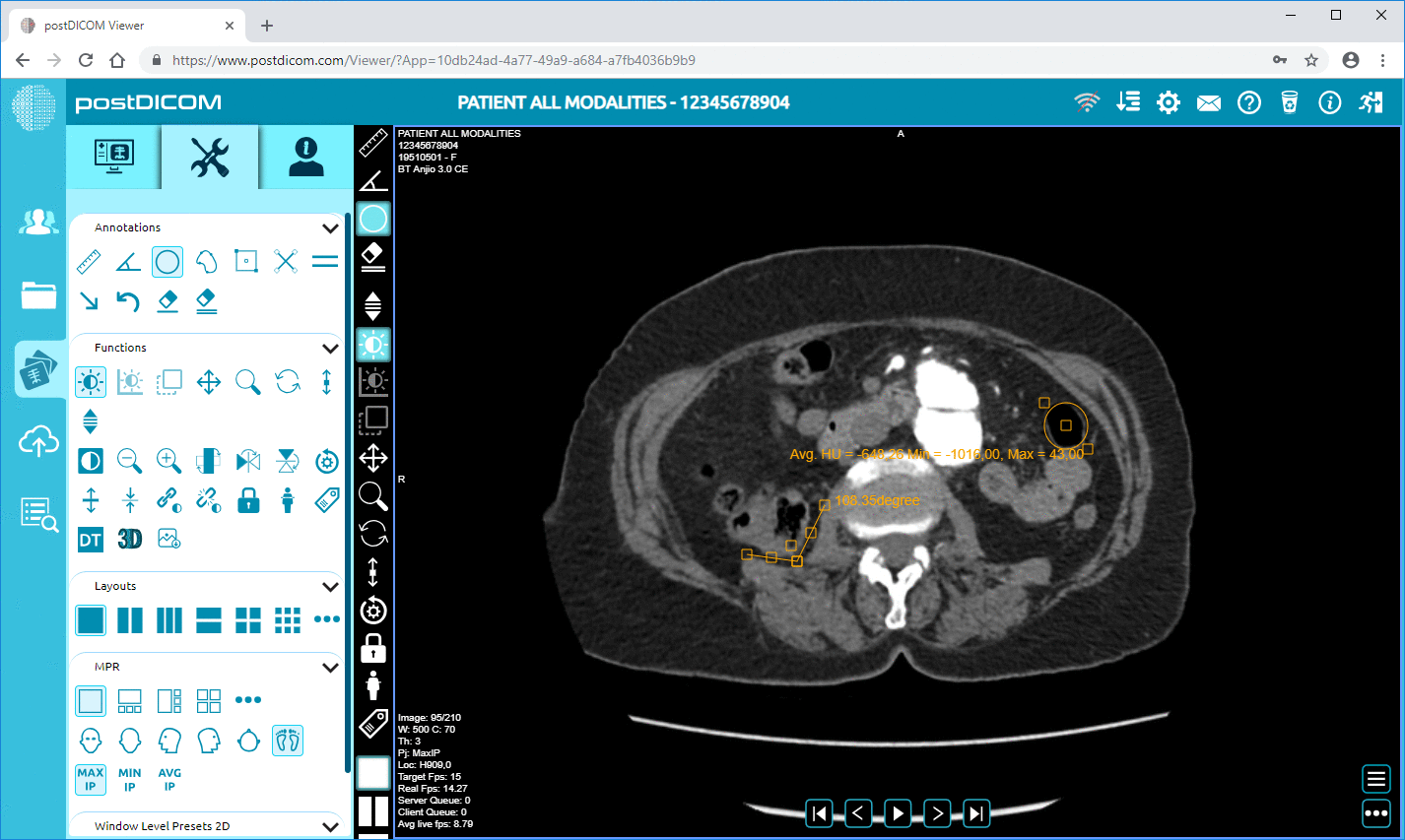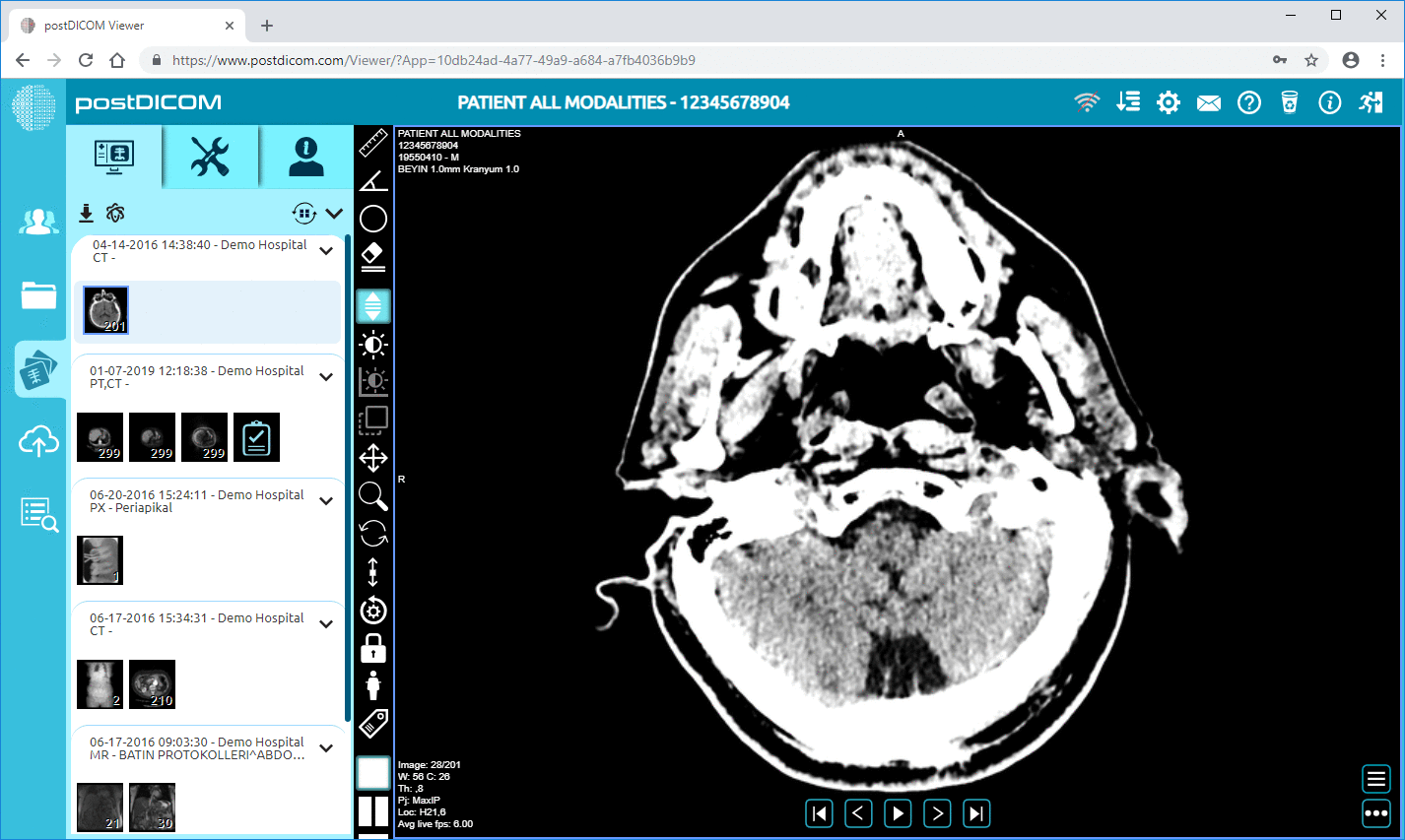
• Avanceret billedbehandling og understøttelse af multimodalitet: Selvom dette vedrører pixels, betyder understøttelsen af modaliteter som PET-CT og multi-serier, at metadataene er betydelige (SUV-værdier, fusionsvolumener, modalitetstype) og tilgængelige til analyse.
Brug af en platform som PostDiCom giver dig mulighed for at udnytte metadata gennem strukturerede arbejdsgange, API'er og skyarkitektur.
Sådan strukturerer du arbejdsgangen fra rå arkiver til analytisk klar indsigt.
 - Created by PostDICOM.jpg)
Det første skridt i at udnytte DICOM-metadata til analyse er ekstraktion og normalisering. Biblioteker som Python-pakken PyDICOM med åben kildekode bruges ofte til at analysere DICOM-filer og udtrække relevante tags, herunder billedrækker og kolonner, konvolutionskerner og modalitetsspecifikke erhvervelsesparametre.
Håndtering af heterogenitet er afgørende, da forskellige leverandører ofte bruger private tags eller ikke-standardiserede implementeringer. Robust parsing, fallback-logik og omfattende tag-mapping tabeller er påkrævet for at sikre konsistens på tværs af datasæt.
Når metadataene er udtrukket, skal de normaliseres og kortlægges til standardontologier og strukturer, såsom modalitetskoder, leverandørnavne, snittykkelseskategorier og standardiserede dato- og tidsformater.
Endelig skal de strukturerede metadata gemmes i et miljø med store data, såsom en relationsdatabase, NoSQL-lager eller kolonneformet datasø, med indeksering for at muliggøre hurtig og effektiv forespørgsel.
Når metadata er udtrukket, skal de gennemgå kvalitetssikring for at sikre nøjagtighed og pålidelighed. Manglende eller inkonsekvente felter, f.eks. tomme snittykkelsesværdier, inkonsekvente modalitetsetiketter eller duplikerede UID'er for studieforekomst, skal identificeres og rettes.
Privatliv og anonymisering er også afgørende på dette tidspunkt, da metadata ofte indeholder personligt identificerbare oplysninger, herunder patientnavne, ID'er, og datoer; afidentifikationsværktøjer og protokoller er vigtige.
Vedligeholdelse af omfattende revisionsspor er en anden vigtig praksis, der dokumenterer, hvornår metadata blev ekstraheret, hvilke parserversioner der blev brugt, og eventuelle fejl, der opstod under processen.
Forvaltningspolitikker bør også definere obligatoriske felter og give vejledning i, hvordan ældre eller ufuldstændige datasæt håndteres for at sikre, at downstream-analyser er nøjagtige og overholder kravene.
Det næste trin er metadata-drevet indeksering og funktionsteknik, som omdanner rå metadata til handlingsbar information.
Dette indebærer oprettelse af indekser og filtre, der giver forskere og analytikere mulighed for at forespørge specifikke datasæt, for eksempel hente alle bryst-CT-scanninger med skivetykkelse under 1,5 millimeter fra en bestemt leverandør inden for et givet datointerval.
Funktionsteknik bygger på dette ved at kombinere metadatafelter såsom leverandør, model, anskaffelsesdato, snittykkelse, konvolutionskerne, kontrastprotokol, kropsregion, strålingsdosis og institutions-id i strukturerede variabler, der er egnede til analyse.
Metadata kan også knyttes til kliniske datasæt, der forbinder billeddata til patientresultater, diagnoser eller behandlinger. Denne forbindelse giver et mere holistisk syn på billeddannelsesdata og dets kliniske kontekst.
Når metadata er indekseret, og funktioner er konstrueret, bliver analyse og indsigtsgenerering mulig.
Beskrivende analyser kan afsløre undersøgelsesmængder efter modalitet, leverandør eller region, spore tendenser i anskaffelsesparametre og fremhæve fejl eller uoverensstemmelser i billedbehandlingspraksis. Sammenlignende analyser muliggør evaluering af erhvervelsesprotokoller på tværs af institutioner, påvisning af afvigelser og identifikation af afvigelser, der kan kræve særlig opmærksomhed.
For maskinlæring og AI-applikationer er metadata afgørende for at kontrollere domæneskift, sikre, at trænings- og testdatasæt stratificeres korrekt, og kombinere pixelbaserede funktioner med strukturerede metadatavariabler. Operationelle dashboards kan derefter udnytte disse data til at overvåge arbejdsbyrden, vurdere kvalitetssikringsmålinger og sikre protokoloverholdelse på tværs af websteder.
Endelig fuldender feedback og løbende forbedringer metadata-livscyklussen. Indsigt, der stammer fra analyser, kan informere om forfining af anskaffelsesprotokoller og standardisering af arbejdsgange for at forbedre den samlede datakvalitet.
Nye billedstudier og metadata bør løbende indtages med overvågning af metadatalagerets ydeevne, forespørgselstider og dataintegritet. Erfaringer bør arkiveres for at fange forudsigelige metadatafelter, afhjælpe tilbagevendende huller eller fejl og forbedre forvaltningspraksis.
Denne iterative tilgang sikrer, at metadatapipeline forbliver robuste, skalerbare og værdifulde til fremtidig forskning, AI-applikationer og operationel beslutningstagning.
• Leverandør-/institutionens variabilitet: Private tags eller løse standardfortolkninger.
• Manglende eller beskadigede metadata: Æl dre undersøgelser kan have ufuldstændige overskrifter.
• Databeskyttelse og anonymisering: PHI skal avidentificeres til forskning på flere steder.
• Skalering og ydeevne: Millioner af billeder kræver effektiv behandling og opbevaring.
• Domæneskift/bias: Dominerende leverandør er/protokoller kan skæve AI-modeller.
• Lovgivnings- og overholdelsesproblemer: Implementeringer i flere regioner kan involvere HIPAA, GDPR eller lokale regler.
DICOM-metadata er det skjulte skelet af billedanalyse. Platforme som PostDICOM illustrerer, hvordan man omdanner et fragmenteret arkiv af DICOM-filer til et søgbart, skalerbart, metadata-drevet økosystem. Hvis du vil udforske PostDiCom, opfordrer vi dig til at få vores 7-dages gratis prøveperiode.
|
Cloud PACS og online DICOM-fremviserUpload DICOM-billeder og kliniske dokumenter til PostDICOM-servere. Gem, se, samarbejd og del dine medicinske billedbehandlingsfiler. |