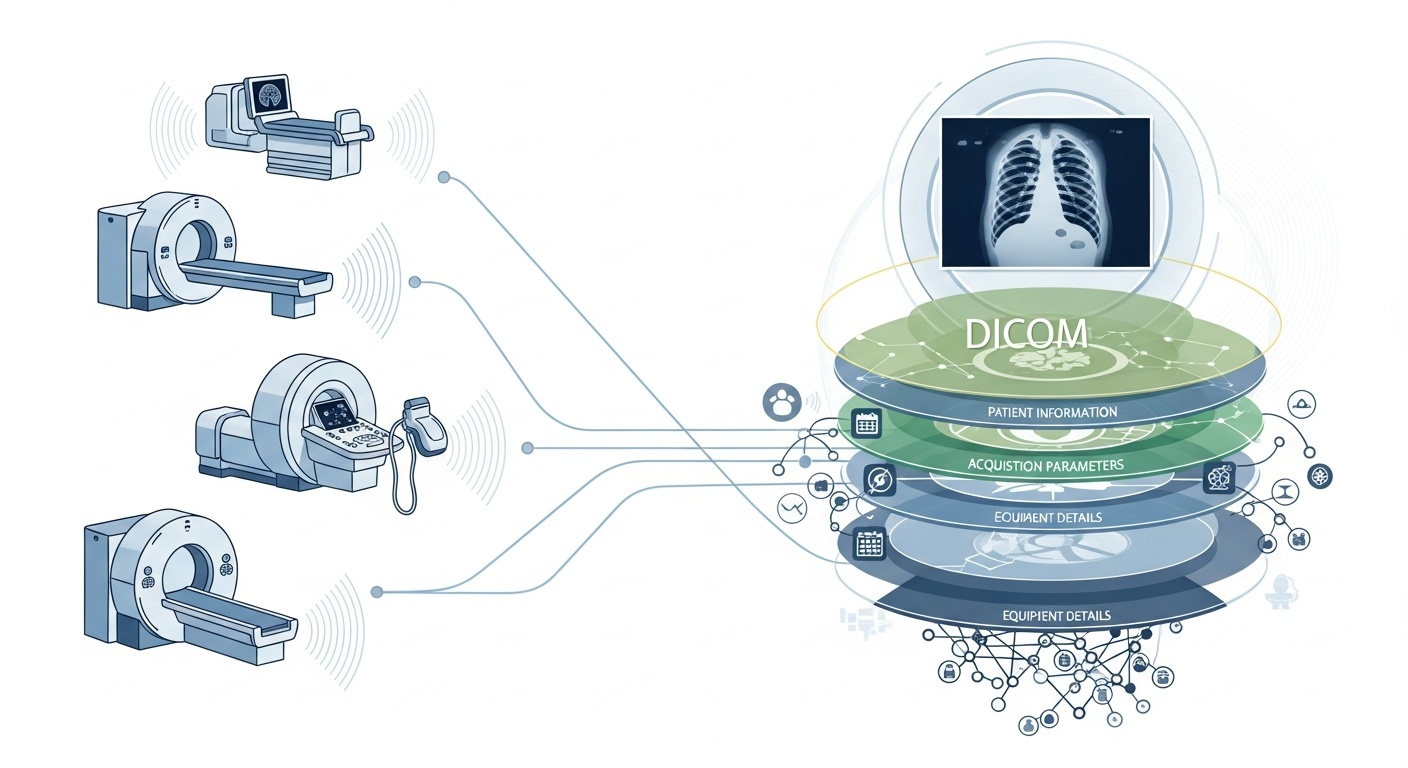
Varje gång ett radiologiteam skannar en patient, säg en CT, MR eller ultraljud, genereras en kaskad av data. Mest synlig är själva bilden, men bakom den finns ett rikt lager av metadata: vem, vad, när, hur och var skanningen. Det lagret styrs av standarden DICOM (Digital Imaging and Communications in Medicine), en medicinsk bildformatstandard som bakades in av National Electrical Manufacturers Association (NEMA) och American College of Radiology (ACR) för decennier sedan.
Det som gör metadata så intressant är att de är strukturerade, maskinläsbara och anmärkningsvärt detaljerade: utrustningsinställningar, anskaffningsparametrar, patientdemografi, studie-ID, till och med institutionskoder och detaljer om tillverkare av modalitet. Den rikedomen är det som möjliggör analys av stora data, nedströms forskning, AI-modellering och protokollstandardisering om du utnyttjar det väl.
I samband med big data talar vi inte bara om några dussin bildstudier. Vi pratar hundratusentals, eller till och med miljontals, bilder över modaliteter, webbplatser och leverantörer, med metadata som nyckelindexeringslager: ”vem & när och hur” för varje objekt. Utan att effektivt använda metadata riskerar du att ha ett massivt arkiv med bilder men minimal förmåga att fråga, jämföra eller få insikt från dem. En ny recension säger: ”majoriteten av informationen som lagras i PACS-arkiv nås aldrig igen”, en bortkastad möjlighet.
Säg att du kör en flerplatsstudie av lung-CT-skanningar för tidig detektering av emfysem. Du vill välja skanningar baserat på parametrar: skannerleverantör/märke/modell, skivtjocklek, rekonstruktionskärna, patientens ålder, datumintervall, kanske till och med dosparametrar. De flesta av dessa är metadatafäl t, inte pixeldata. Genom att extrahera dessa taggar kan du bygga kohorten, utesluta inkompatibla skanningar (t.ex. för tjocka segment) och säkerställa jämförbarhet.
Metadata låter dig övervaka själva bildprocessen: använder institutionen rätt protokoll? Avviker förvärvsparametrar över tiden (t.ex. synfält, kontrastinjektionstid)? Är leverantörsinställningarna konsekventa? I en stordatavärld där tusentals skanningar sker per dag kan du inte lita på mänsklig ögonboll. Du behöver analys på metadata. Många PACS-system underutnyttjar metadata av denna anledning.
Om du bygger en AI- eller radiomics-pipeline kan du inte behandla varje bild som utbytbar. Metadata blir en integrerad kontrollvariabel: inmatningsfunktioner inkluderar ofta modalitet, kVP (kilovoltagepak), tillverkare, kärna, skivtjocklek och till och med datum eller sjukhus kan betyda (domänskift). Dessa metadatafält hjälper till att hantera partiskhet, harmonisera data och kommentera bilder med sammanhang. Många forskare kallar metadata ”lika viktigt som pixeldata.”
Big data betyder skala. Det innebär olika källor, flera leverantörer, olika institutioner och heterogena format. DICOM-metadata är det standardiserade ”språket” som hjälper till att förena metadatalagret, möjliggöra sökning/indexering, säkerställa interoperabilitet och bygga skalbara arkitekturer (moln-PACs, federerade arkiv). Men genomförandet är viktigt. Samma PMC-studie fann att många system inte utnyttjar standarden fullt ut.
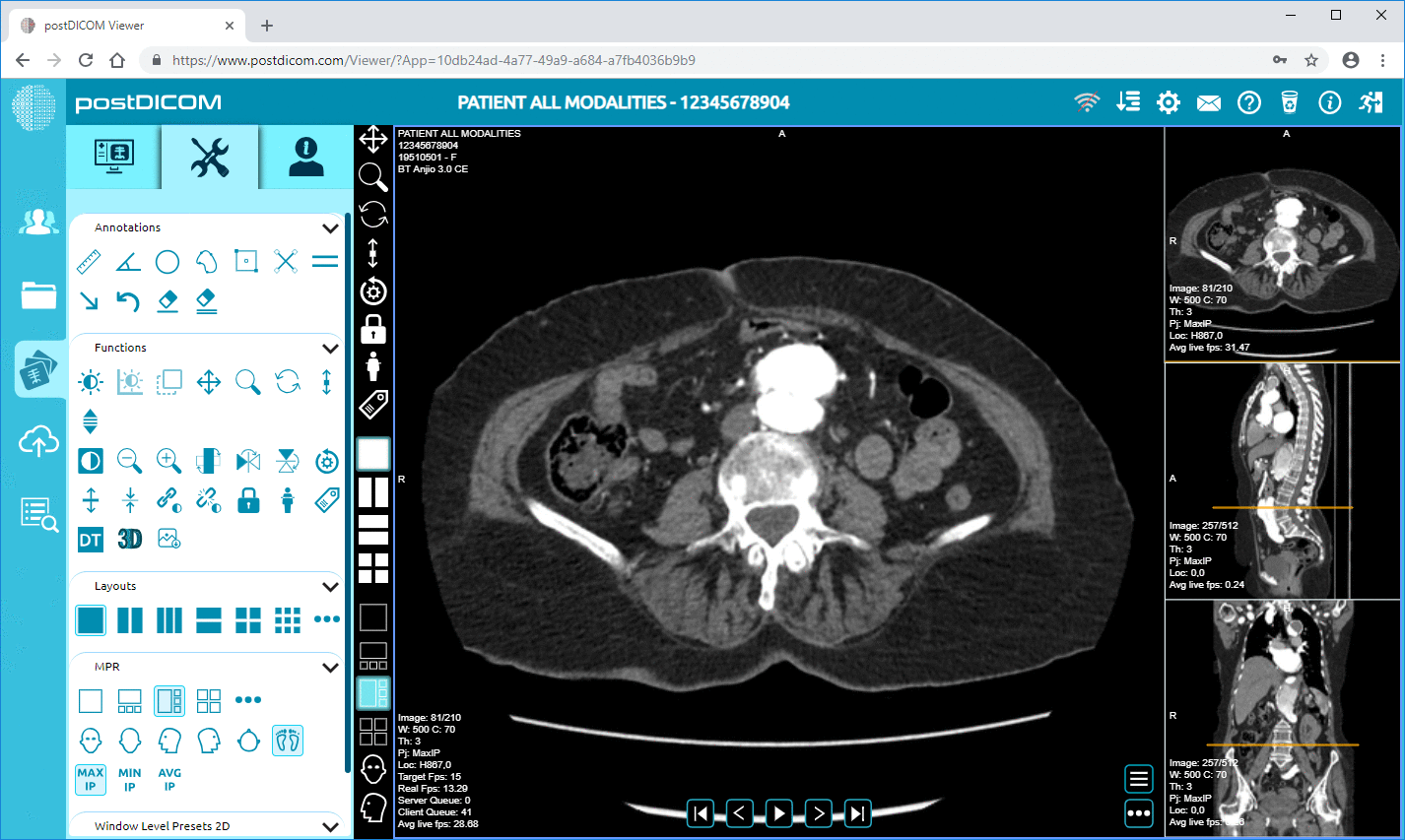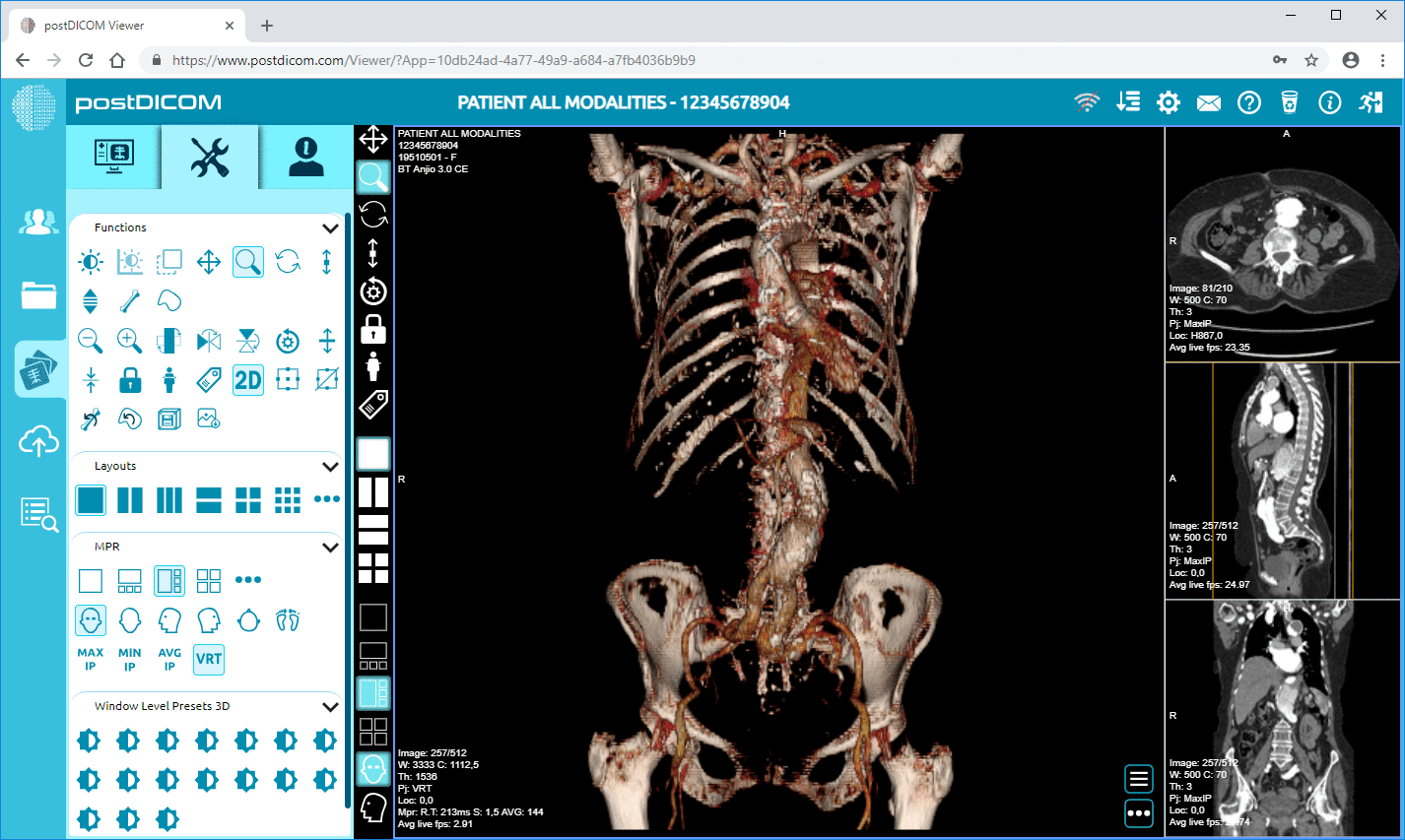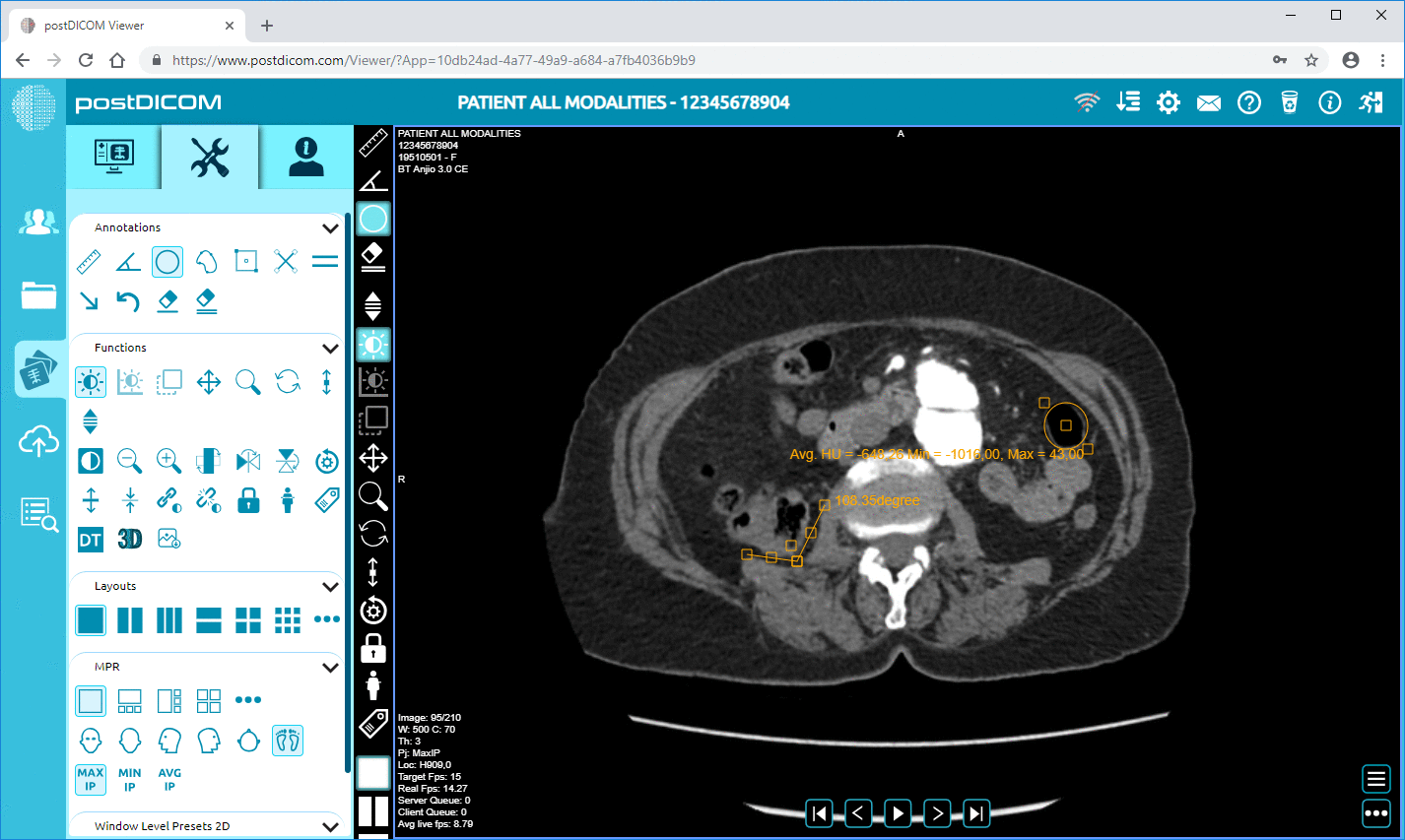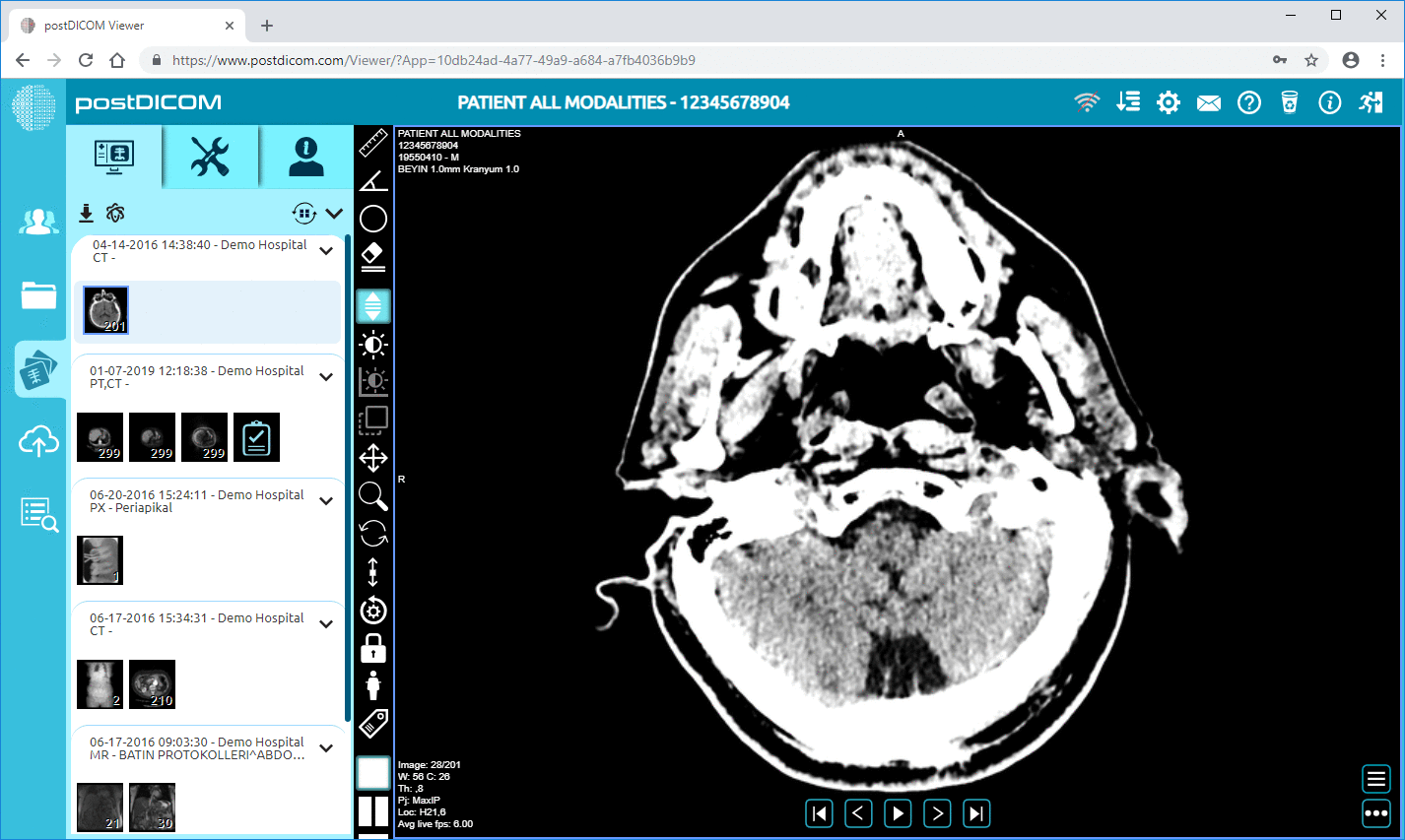
PostDiCom erbjuder ett molnbaserat PACS (Picture Archiving and Communication System) för lagring, visning och delning av bildstudier och kliniska dokument. Några viktiga metadata-relevanta funktioner i PostDiCom:
• Stöd för Dicom-taggar och beskrivningar: Vårt resursbibliotek listar ”DICOM Modality & Tags”, så att användare kan komma åt tagglistor och beskrivningar. DICOM-modalitet och taggar
• Api/Fhir-integration: Den stöder API- och FHIR-gränssnitt (Fast Healthcare Interoperability Resources), vilket gör att metadata kan frågas programmatiskt, integreras med andra system och analyseras.
• Molnskalbarhet och delning på flera platser: Delning mellan patienter, läkare, institutioner; obegränsad skalbarhet innebär att stora datapipeliner blir genomförbara.
• Avancerad bildbehandling och multimodalitetsstöd: Även om detta gäller pixlar, innebär stödet för modaliteter som PET-CT och multiserier att metadata är betydande (SUV-värden, fusionsvolymer, modalitetstyp) och tillgängliga för analys.
Genom att använda en plattform som PostDiCom kan du utnyttja metadata genom strukturerade arbetsflöden, API: er och molnarkitektur.
Så här strukturerar du arbetsflödet från råa arkiv till analytiska insikter.
 - Created by PostDICOM.jpg)
Det första steget i att utnyttja DICOM-metadata för analys är extraktion och normalisering. Bibliotek som Python-paketet PyDICOM med öppen källkod används ofta för att analysera DICOM-filer och extrahera relevanta taggar, inklusive bildrader och kolumner, konvolutionskärnor och modalitetsspecifika förvärvsparametrar.
Att hantera heterogenitet är avgörande, eftersom olika leverantörer ofta använder privata taggar eller icke-standardiserade implementeringar. Robust analys, fallback-logik och omfattande tag-mappningstabeller krävs för att säkerställa konsistens mellan datamängder.
När metadata har extraherats måste de normaliseras och mappas till standardontologier och strukturer, såsom modalitetskoder, leverantörsnamn, segmenttjocklekskategorier och standardiserade datum- och tidsformat.
Slutligen bör de strukturerade metadata lagras i en stor datamiljö, till exempel en relationsdatabas, NoSQL-butik eller kolumnar datasjö, med indexering för att möjliggöra snabb och effektiv förfrågan.
När metadata har extraherats måste de genomgå kvalitetssäkring för att säkerställa noggrannhet och tillförlitlighet. Fält som saknas eller är inkonsekventa, till exempel tomma snitttjockleksvärden, inkonsekventa modalitetsetiketter eller dubbla UID för studieinstanser, måste identifieras och korrigeras.
Sekretess och anonymisering är också avgörande i detta skede, eftersom metadata ofta innehåller personligt identifierbar information inklusive patientnamn, ID, och datum; avidentifieringsverktyg och protokoll är viktiga.
Att upprätthålla omfattande granskningsspår är en annan viktig praxis, dokumentera när metadata extraherades, vilka parserversioner som användes och eventuella fel som uppstod under processen.
Styrningspolicyer bör också definiera obligatoriska fält och ge vägledning om hur äldre eller ofullständiga datamängder ska hanteras för att säkerställa att nedströmsanalyser är korrekta och överensstämmer med kraven.
Nästa steg är metadatadriven indexering och funktionsteknik, som omvandlar råa metadata till handlingsbar information.
Detta innebär att skapa index och filter som gör det möjligt för forskare och analytiker att fråga specifika datamängder, till exempel hämta alla bröstCT-skanningar med skivtjocklek under 1,5 millimeter från en viss leverantör inom ett visst datumintervall.
Funktionsteknik bygger på detta genom att kombinera metadatafält som leverantör, modell, förvärvsdatum, skivtjocklek, konvolutionskärna, kontrastprotokoll, kroppsregion, strålningsdos, och institutions-ID till strukturerade variabler som är lämpliga för analys.
Metadata kan också kopplas till kliniska datamängder, koppla bilddata till patientresultat, diagnoser eller behandlingar. Denna koppling möjliggör en mer holistisk syn på bilddata och dess kliniska sammanhang.
När metadata har indexerats och funktioner konstruerats blir analys och insiktsgenerering möjlig.
Beskrivande analys kan avslöja studievolymer efter modalitet, leverantör eller region, spåra trender i förvärvsparametrar och markera fel eller inkonsekvenser i bildbehandlingsmetoder. Jämförande analys möjliggör utvärdering av förvärvsprotokoll över institutioner, upptäckt av avvikelser och identifiering av avvikande skanningar som kan kräva särskild uppmärksamhet.
För maskininlärning och AI-applikationer är metadata avgörande för att kontrollera domänskift, se till att utbildnings- och testdatauppsättningar stratifieras på lämpligt sätt och kombinera pixelbaserade funktioner med strukturerade metadatavariabler. Operativa instrumentpaneler kan sedan utnyttja dessa data för att övervaka arbetsbelastningen, bedöma kvalitetssäkringsmått och säkerställa protokollöverensstämmelse på alla webbplatser.
Slutligen kompletterar feedback och kontinuerlig förbättring metadatans livscykel. Insikter som härrör från analys kan informera om förfining av anskaffningsprotokoll och standardisering av arbetsflöden för att förbättra den övergripande datakvaliteten.
Nya bildstudier och metadata bör kontinuerligt tas in, med övervakning av metadatalagrens prestanda, frågetider och dataintegritet. Lärdomar bör arkiveras för att fånga förutsägbara metadatafält, åtgärda återkommande luckor eller fel och förbättra förvaltningspraxis.
Detta iterativa tillvägagångssätt säkerställer att metadata pipeline förblir robusta, skalbara och värdefulla för framtida forskning, AI-applikationer och operativt beslutsfattande.
• Leverantör/institution Variabilitet: Privata taggar eller lösa standardtolkningar.
• Saknade eller skadade metadata: Äl dre studier kan ha ofullständiga rubriker.
• Datasekretess och anonymisering: PHI måste avidentifieras för forskning på flera platser.
• Skala och prestanda: Miljontals bilder kräver effektiv bearbetning och lagring.
• Domänskift/bias: Dominanta leverantörer/protokoll kan snedvrida AI-modeller.
• Regler- och efterlevnadsproblem: Distributioner i flera regioner kan involvera HIPAA, GDPR eller lokala bestämmelser.
DICOM-metadata är det dolda skelettet för bildanalys. Plattformar som PostDICOM illustrerar hur man omvandlar ett fragmenterat arkiv med DICOM-filer till ett sökbart, skalbart, metadatadrivet ekosystem. Om du vill utforska PostDiCom uppmuntrar vi dig att få vår 7-dag ars gratis provperiod.
|
Cloud PACS och DICOM-visare onlineLadda upp DICOM-bilder och kliniska dokument till PostDICOM-servrar. Lagra, visa, samarbeta och dela dina medicinska bildfiler. |