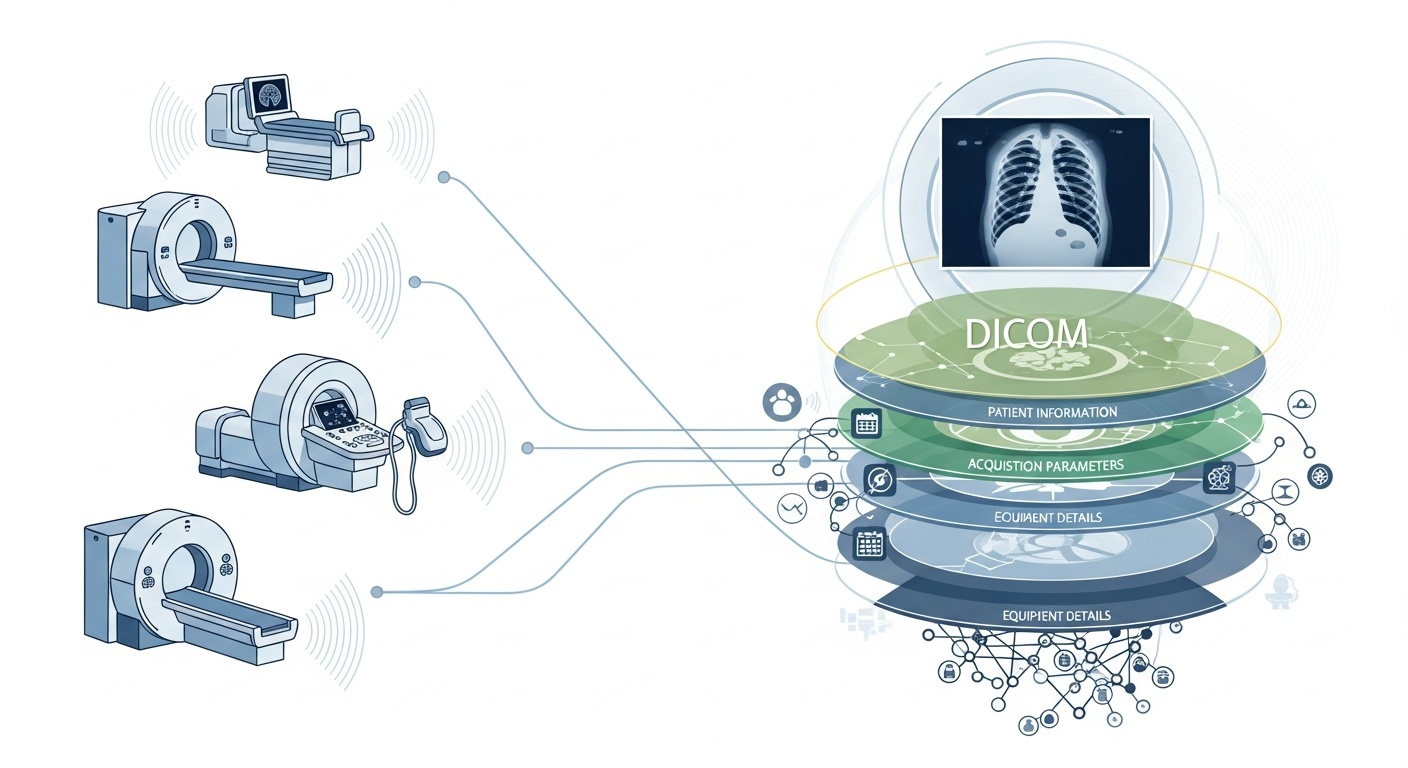
Elke keer dat een radiologieteam een patiënt scant, bijvoorbeeld een CT, MRI of echografie, wordt er een stroom aan gegevens gegenereerd. Het meest zichtbaar is het beeld zelf, maar daarachter schuilt een rijke laag metadata: wie, wat, wanneer, hoe en waar van de scan. Die laag wordt beheerst door de standaard DICOM (Digital Imaging and Communications in Medicine), een standaard voor medische beeldformaten die tientallen jaren geleden is opgesteld door de National Electrical Manufacturers Association (NEMA) en het American College of Radiology (ACR).
Wat metadata zo interessant maakt, is dat deze gestructureerd, machineleesbaar en opmerkelijk gedetailleerd is: apparatuurinstellingen, acquisitieparameters, patiëntdemografie, studie-ID's, zelfs instellingscodes en gegevens van de modaliteitsfabrikant. Die rijkdom maakt big data-analyse, vervolgonderzoek, AI-modellering en protocolstandaardisatie mogelijk als u deze goed benut.
In de context van big data hebben we het niet slechts over enkele tientallen beeldvormende onderzoeken. We hebben het over honderdduizenden of zelfs miljoenen beelden over verschillende modaliteiten, locaties en leveranciers heen, met metadata als de belangrijkste indexeringslaag: de 'wie & wanneer & hoe' van elk item. Zonder metadata effectief te gebruiken, riskeert u een enorm archief aan beelden te hebben met minimale mogelijkheden om deze te doorzoeken, te vergelijken of er inzichten uit te halen. Een recente recensie stelt: "het merendeel van de informatie die in PACS-archieven is opgeslagen, wordt nooit meer geraadpleegd", een gemiste kans.
Stel dat u een onderzoek op meerdere locaties uitvoert naar long-CT-scans voor de vroege detectie van emfyseem. U wilt scans selecteren op basis van parameters: leverancier/merk/model van de scanner, snededikte, reconstructiekernel, leeftijd van de patiënt, datumbereik, misschien zelfs dosisparameters. De meeste daarvan zijn metadatavelden, geen pixelgegevens. Het extraheren van die tags stelt u in staat om het cohort op te bouwen, incompatibele scans uit te sluiten (bijv. te dikke sneden) en vergelijkbaarheid te garanderen.
Met metadata kunt u het beeldvormingsproces zelf bewaken: gebruikt de instelling het juiste protocol? Wijken acquisitieparameters in de loop van de tijd af (bijv. gezichtsveld, timing van contrastinjectie)? Zijn de instellingen van de leverancier consistent? In een big data-wereld waar duizenden scans per dag plaatsvinden, kunt u niet vertrouwen op visuele controle door mensen. U heeft analyses op metadata nodig. Veel PACS-systemen benutten metadata om deze reden onvoldoende.
Als u een AI- of radiomics-pipeline bouwt, kunt u niet elk beeld als uitwisselbaar beschouwen. Metadata wordt een integrale controlevariabele: invoerkenmerken omvatten vaak modaliteit, kVp (kilovoltage piek), fabrikant, kernel, snededikte en zelfs de datum of het ziekenhuis kunnen van belang zijn (domeinverschuiving). Deze metadatavelden helpen bias te beheren, gegevens te harmoniseren en beelden van context te voorzien. Veel onderzoekers noemen metadata "net zo belangrijk als pixelgegevens".
Big data betekent schaal. Dat impliceert gevarieerde bronnen, meerdere leveranciers, verschillende instellingen en heterogene formaten. DICOM-metadata is de gestandaardiseerde "taal" die helpt de metadatalaag te verenigen, zoeken/indexeren mogelijk te maken, interoperabiliteit te garanderen en schaalbare architecturen te bouwen (cloud-PACS, gefedereerde archieven). Maar implementatie is belangrijk. Dezelfde PMC-studie wees uit dat veel systemen de standaard niet volledig benutten.
PostDICOM biedt een cloudgebaseerd PACS (Picture Archiving and Communication System) voor het opslaan, bekijken en delen van beeldvormende studies en klinische documenten. Enkele belangrijke metadata-relevante functies van PostDICOM:
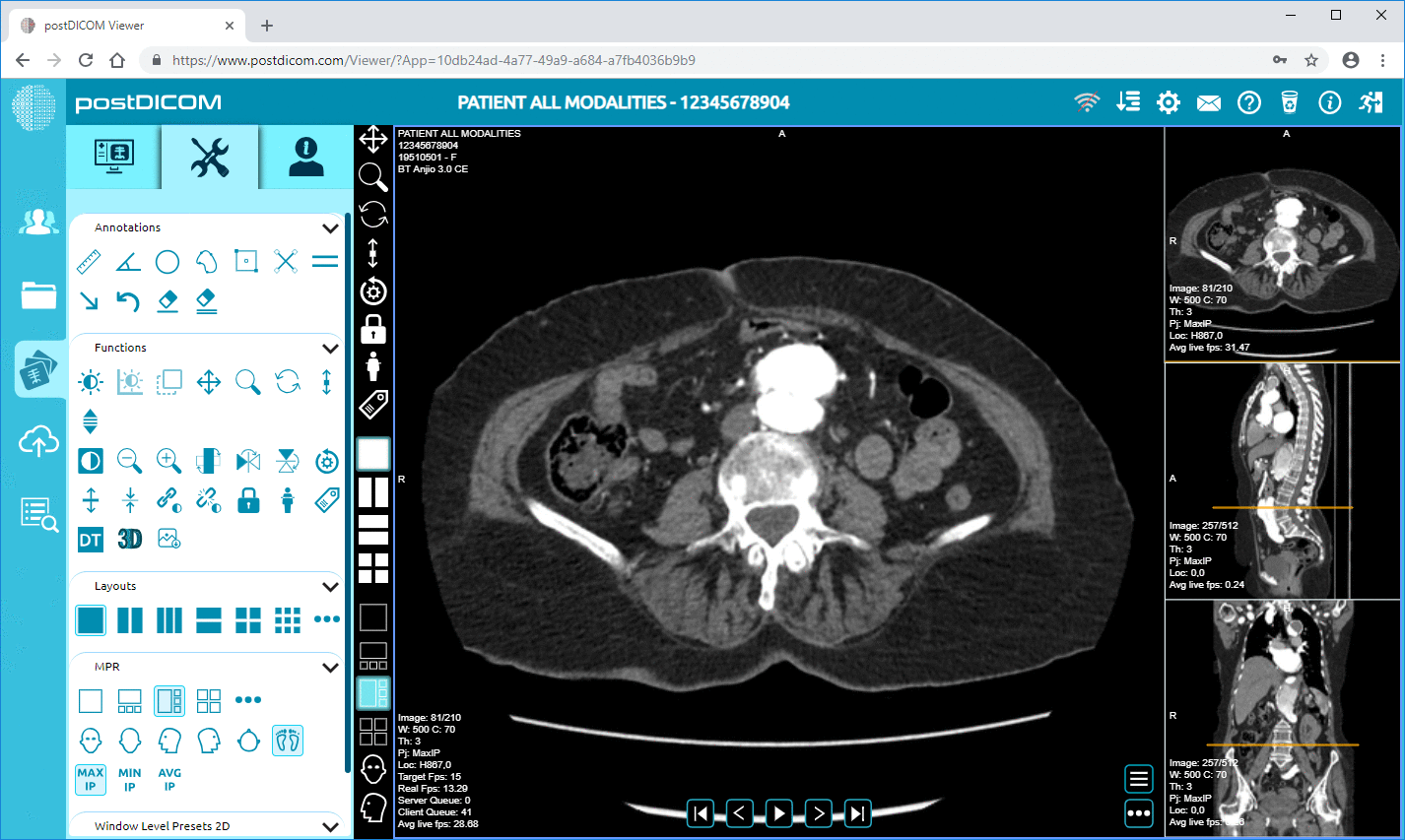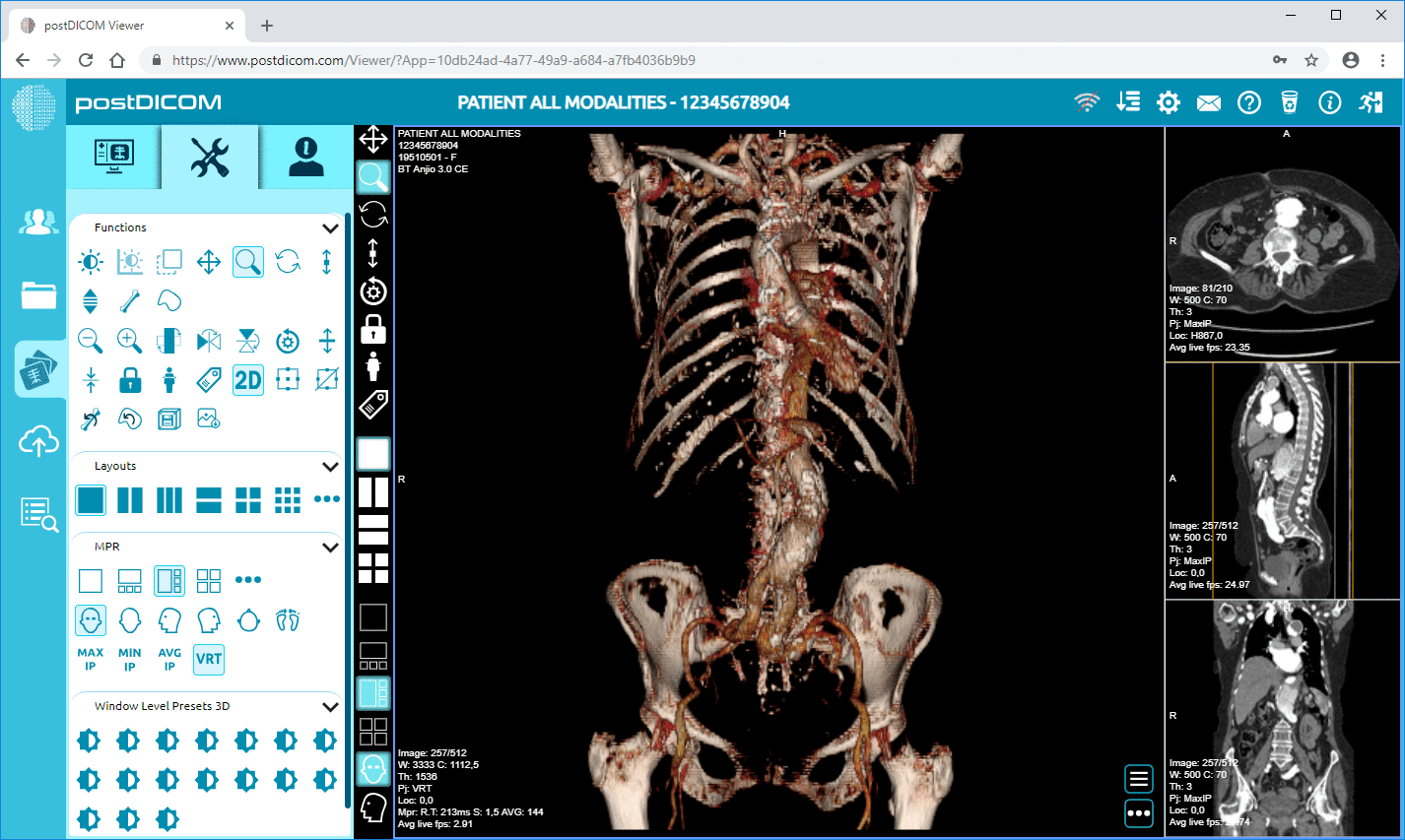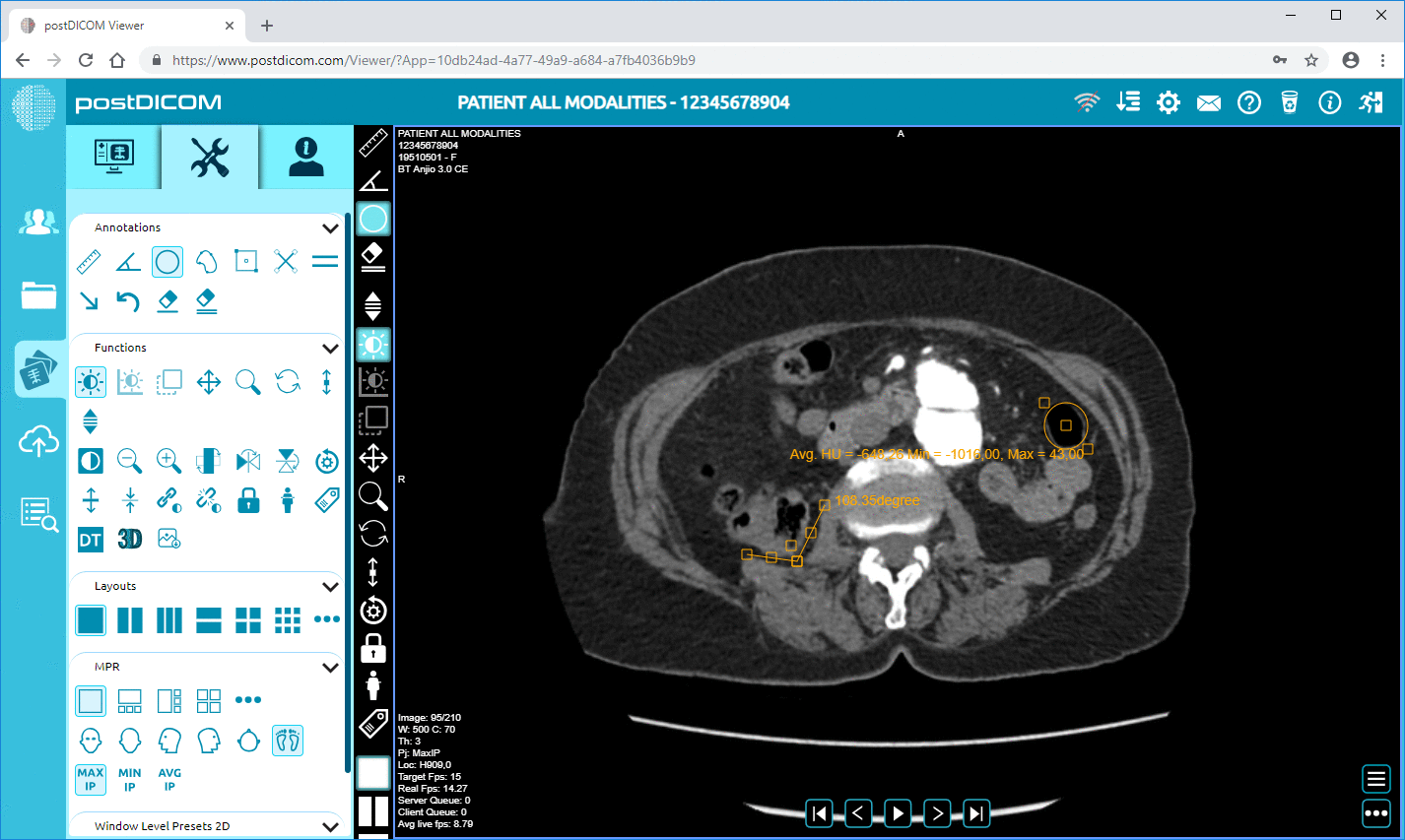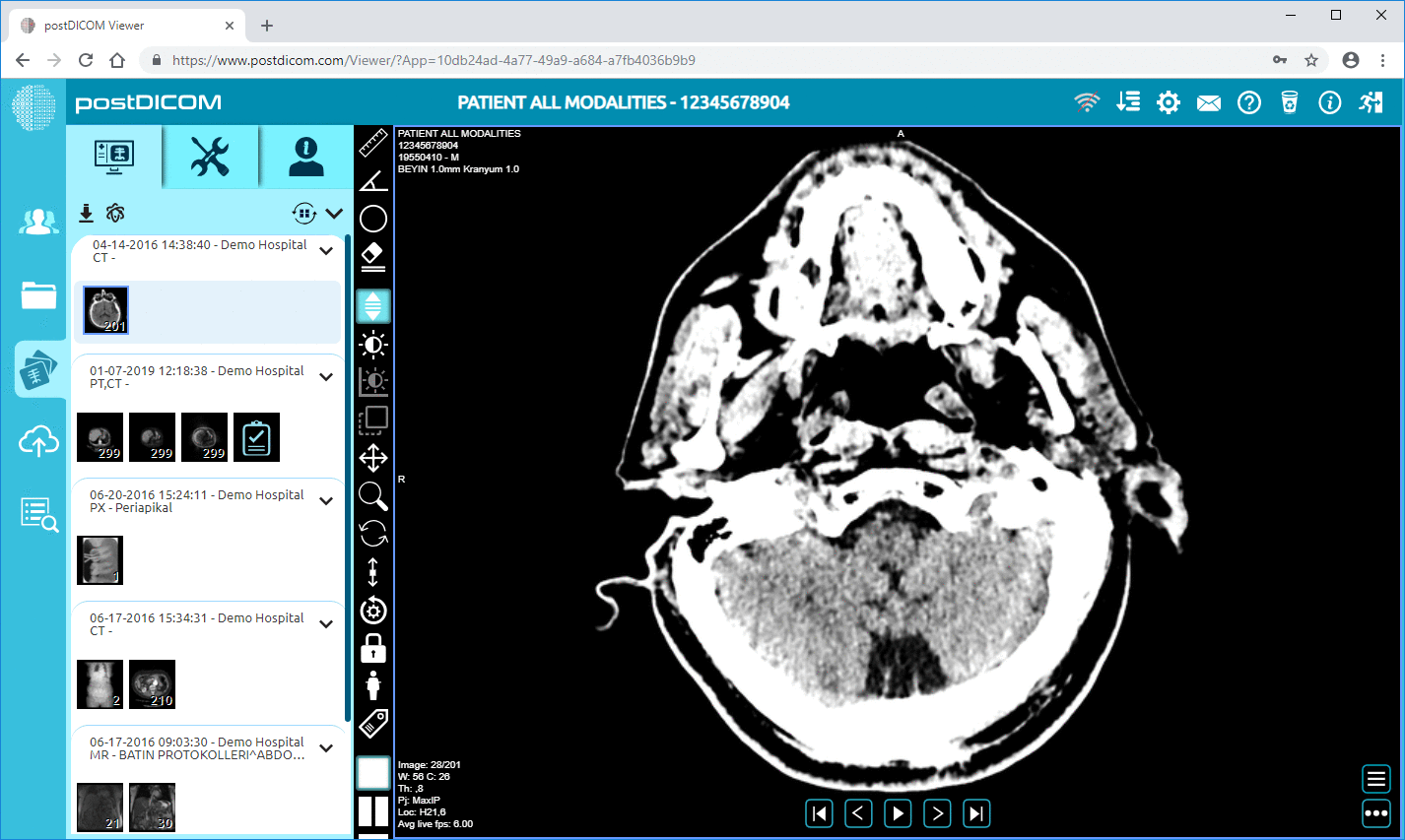
• Ondersteuning voor DICOM-tags & beschrijvingen: Onze bronnenbibliotheek vermeldt "DICOM-modaliteit & Tags", waarmee gebruikers toegang krijgen tot taglijsten en beschrijvingen.DICOM-modaliteit & Tags
• API / FHIR-integratie: Het ondersteunt API- en FHIR-interfaces (Fast Healthcare Interoperability Resources), waardoor metadata programmatisch kan worden opgevraagd, geïntegreerd met andere systemen en geanalyseerd.
• Cloudschaalbaarheid & delen over meerdere locaties: Delen tussen patiënten, artsen, instellingen; onbeperkte schaalbaarheid betekent dat big data-pipelines haalbaar worden.
• Geavanceerde beeldverwerking & multimodale ondersteuning: Hoewel dit pixels betreft, betekent de ondersteuning van modaliteiten zoals PET–CT en multi-series dat de metadata substantieel is (SUV-waarden, fusievolumes, modaliteitstype) en beschikbaar is voor analyse.
Met een platform zoals PostDICOM kunt u metadata benutten via gestructureerde workflows, API's en cloudarchitectuur.
Hier is hoe u de workflow structureert van ruwe archieven tot inzichten die klaar zijn voor analyse.
 - Created by PostDICOM.jpg)
De eerste stap in het benutten van DICOM-metadata voor analyse is extractie en normalisatie. Bibliotheken zoals het open-source Python-pakket PyDicom worden vaak gebruikt om DICOM-bestanden te parsen en relevante tags te extraheren, waaronder beeldrijen en -kolommen, convolutiekernels en modaliteitsspecifieke acquisitieparameters.
Het omgaan met heterogeniteit is cruciaal, aangezien verschillende leveranciers vaak privétags of niet-standaard implementaties gebruiken. Robuuste parsing, fallback-logica en uitgebreide tag-mappingtabellen zijn vereist om consistentie tussen datasets te garanderen.
Eenmaal geëxtraheerd, moet de metadata worden genormaliseerd en gekoppeld aan standaard ontologieën en structuren, zoals modaliteitscodes, namen van leveranciers, categorieën voor snededikte en gestandaardiseerde datum- en tijdnotaties.
Ten slotte moet de gestructureerde metadata worden opgeslagen in een big data-omgeving, zoals een relationele database, NoSQL-opslag of kolomgebaseerd datameer, met indexering om snelle, efficiënte bevraging mogelijk te maken.
Eenmaal geëxtraheerd, moet metadata een kwaliteitsborging ondergaan om nauwkeurigheid en betrouwbaarheid te garanderen. Ontbrekende of inconsistente velden, zoals lege waarden voor snededikte, inconsistente modaliteitslabels of dubbele Study Instance UID's, moeten worden geïdentificeerd en gecorrigeerd.
Privacy en anonimisering zijn ook in dit stadium van cruciaal belang, aangezien metadata vaak persoonlijk identificeerbare informatie bevat, waaronder namen van patiënten, ID's en data; de-identificatietools en -protocollen zijn essentieel.
Het bijhouden van uitgebreide audit trails is een andere belangrijke praktijk, waarbij wordt gedocumenteerd wanneer metadata is geëxtraheerd, welke parserversies zijn gebruikt en eventuele fouten die tijdens het proces zijn opgetreden.
Beheersbeleid moet ook verplichte velden definiëren en richtlijnen bieden voor het omgaan met verouderde of onvolledige datasets om ervoor te zorgen dat vervolganalyses nauwkeurig en conform de regels zijn.
De volgende stap is metadata-gestuurde indexering en feature engineering, waarbij ruwe metadata wordt omgezet in bruikbare informatie.
Dit omvat het creëren van indexen en filters waarmee onderzoekers en analisten specifieke datasets kunnen opvragen, bijvoorbeeld het ophalen van alle thorax-CT-scans met een snededikte van minder dan 1,5 millimeter van een bepaalde leverancier binnen een bepaald datumbereik.
Feature engineering bouwt hierop voort door metadatavelden zoals leverancier, model, acquisitiedatum, snededikte, convolutiekernel, contrastprotocol, lichaamsregio, stralingsdosis en instellings-ID te combineren tot gestructureerde variabelen die geschikt zijn voor analyse.
Metadata kan ook worden gekoppeld aan klinische datasets, waarbij beeldvormende gegevens worden verbonden met patiëntuitkomsten, diagnoses of behandelingen. Deze koppeling maakt een meer holistisch beeld van beeldvormende gegevens en hun klinische context mogelijk.
Zodra metadata is geïndexeerd en functies zijn ontwikkeld, worden analyses en het genereren van inzichten mogelijk.
Beschrijvende analyses kunnen studievolumes per modaliteit, leverancier of regio onthullen, trends in acquisitieparameters volgen en fouten of inconsistenties in beeldvormingspraktijken benadrukken. Vergelijkende analyses maken evaluatie van acquisitieprotocollen tussen instellingen mogelijk, detectie van afwijkingen en identificatie van uitschieters die speciale aandacht vereisen.
Voor machine learning- en AI-toepassingen is metadata essentieel voor het beheersen van domeinverschuiving, om ervoor te zorgen dat trainings- en testdatasets op de juiste manier worden gestratificeerd en om op pixels gebaseerde kenmerken te combineren met gestructureerde metadatavariabelen. Operationele dashboards kunnen deze gegevens vervolgens gebruiken om de werkbelasting te bewaken, metrics voor kwaliteitsborging te beoordelen en protocolnaleving op verschillende locaties te waarborgen.
Ten slotte voltooien feedback en continue verbetering de levenscyclus van metadata. Inzichten verkregen uit analyses kunnen de verfijning van acquisitieprotocollen en standaardisatie van workflows informeren om de algehele datakwaliteit te verbeteren.
Nieuwe beeldvormende studies en metadata moeten continu worden opgenomen, met bewaking van de prestaties van de metadata-opslag, querytijden en gegevensintegriteit. Geleerde lessen moeten worden gearchiveerd om voorspellende metadatavelden vast te leggen, terugkerende hiaten of fouten aan te pakken en beheerpraktijken te verbeteren.
Deze iteratieve aanpak zorgt ervoor dat metadata-pipelines robuust, schaalbaar en waardevol blijven voor toekomstig onderzoek, AI-toepassingen en operationele besluitvorming.
• Variabiliteit leverancier/instelling: Privétags of losse interpretaties van de standaard.
• Ontbrekende of beschadigde metadata: Oudere studies hebben mogelijk onvolledige headers.
• Gegevensprivacy & anonimisering: PHI moet worden gede-identificeerd voor onderzoek op meerdere locaties.
• Schaal & prestaties: Miljoenen beelden vereisen efficiënte verwerking en opslag.
• Domeinverschuiving/bias: Dominante leveranciers/protocollen kunnen AI-modellen vertekenen.
• Regelgevings- & nalevingskwesties: Implementaties in meerdere regio's kunnen te maken krijgen met HIPAA, AVG (GDPR) of lokale regelgeving.
DICOM-metadata is het verborgen skelet van beeldvormingsanalyse. Platforms zoals PostDICOM illustreren hoe een gefragmenteerd archief van DICOM-bestanden kan worden omgezet in een doorzoekbaar, schaalbaar, metadata-gestuurd ecosysteem. Als u PostDICOM wilt verkennen, moedigen we u aan om onze gratis proefperiode van 7 dagen te nemen.
|
Cloud PACS en online DICOM-viewerUpload DICOM-beelden en klinische documenten naar PostDICOM-servers. Sla uw medische beeldbestanden op, bekijk ze, werk samen en deel ze. |