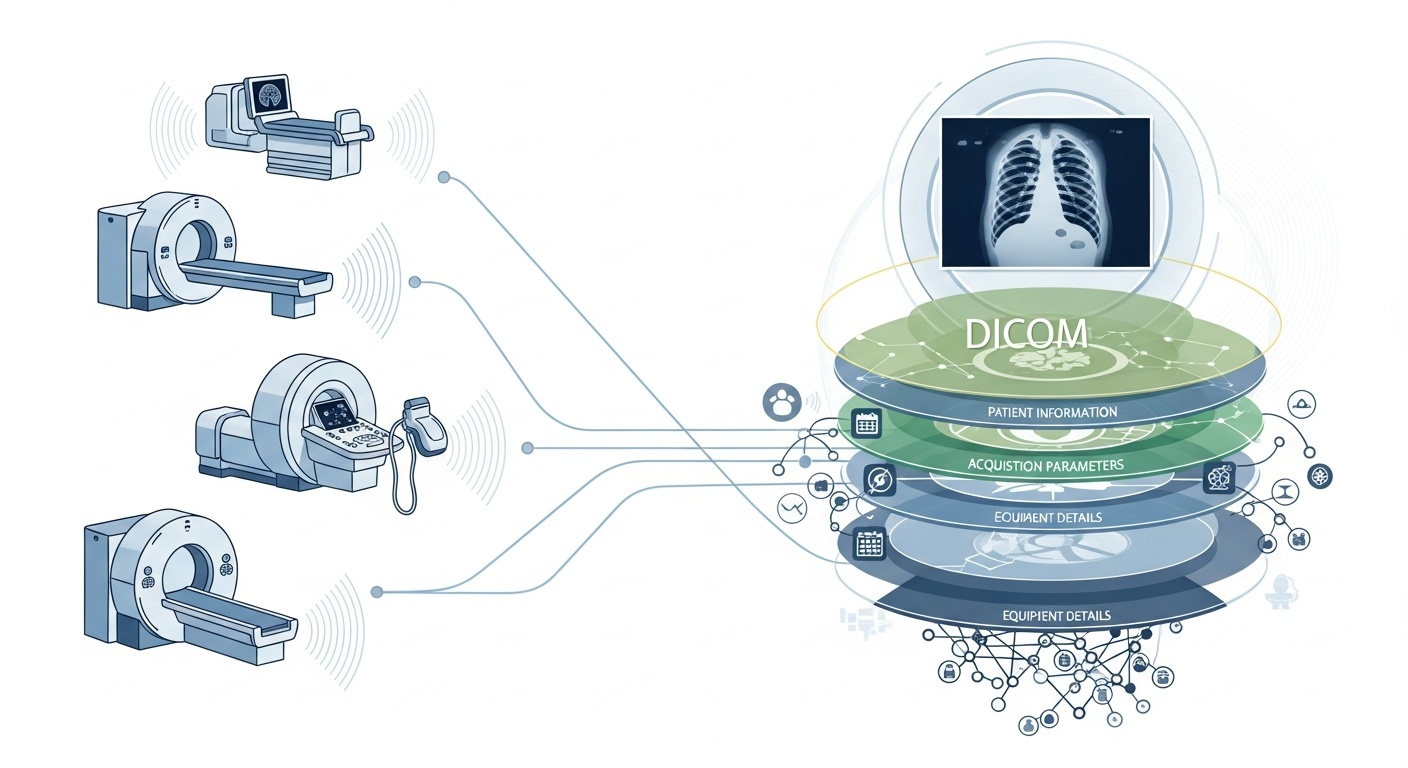
Jedes Mal, wenn ein Radiologieteam einen Patienten scannt, beispielsweise ein CT, MRT oder Ultraschall, wird eine Kaskade von Daten generiert. Am sichtbarsten ist das Bild selbst, aber dahinter verbirgt sich eine Vielzahl von Metadaten: wer, was, wann, wie und wo des Scans. Diese Ebene unterliegt dem Standard DICOM (Digital Imaging and Communications in Medicine), einem Standard für medizinische Bildgebung, der vor Jahrzehnten von der National Electrical Manufacturers Association (NEMA) und dem American College of Radiology (ACR) eingeführt wurde.
Was die Metadaten so interessant macht, ist, dass sie strukturiert, maschinenlesbar und bemerkenswert detailliert sind: Geräteeinstellungen, Erfassungsparameter, Patientendemografie, Studien-IDs, sogar Institutionscodes und Angaben zum Hersteller der Modalität. Dieser Reichtum ermöglicht Big-Data-Analysen, nachgelagerte Forschung, KI-Modellierung und Protokollstandardisierung, wenn man sie richtig nutzt.
Im Zusammenhang mit Big Data sprechen wir nicht nur von ein paar Dutzend bildgebenden Studien. Wir sprechen von Hunderttausenden oder sogar Millionen von Bildern verschiedener Modalitäten, Websites und Anbieter, wobei Metadaten die wichtigste Indexierungsebene sind: das „Wer, wann und wie“ der einzelnen Elemente. Ohne effektive Nutzung von Metadaten riskieren Sie, ein riesiges Bildarchiv zu haben, aber nur minimale Möglichkeiten, diese abzufragen, zu vergleichen oder Erkenntnisse daraus abzuleiten. In einer kürzlich veröffentlichten Studie heißt es: „Auf die Mehrheit der in PACS-Archiven gespeicherten Informationen wird nie wieder zugegriffen“ — eine verpasste Gelegenheit.
Angenommen, Sie führen eine standortübergreifende Studie mit Lungen-CT-Scans zur Früherkennung von Emphysemen durch. Sie sollten die Scans anhand von Parametern auswählen: Hersteller/Marke/Modell des Scanners, Schichtdicke, Rekonstruktionskern, Alter des Patienten, Datumsbereiche, vielleicht sogar Dosisparameter. Die meisten davon sind Metadatenfelder, keine Pixeldaten. Durch Extrahieren dieser Tags können Sie die Kohorte erstellen, inkompatible Scans (z. B. zu dicke Scheiben) ausschließen und die Vergleichbarkeit sicherstellen.
Mithilfe von Metadaten können Sie den Bildgebungsprozess selbst überwachen: Verwendet die Institution das richtige Protokoll? Weichen die Erfassungsparameter im Laufe der Zeit ab (z. B. Sichtfeld, Zeitpunkt der Kontrasteinspritzung)? Sind die Einstellungen des Anbieters konsistent? In einer Welt voller großer Datenmengen, in der täglich Tausende von Scans durchgeführt werden, können Sie sich nicht darauf verlassen, dass die Augen von Menschen beobachtet werden. Sie benötigen Analysen zu Metadaten. Viele PACS-Systeme nutzen Metadaten aus diesem Grund nicht ausreichend aus.
Wenn Sie eine KI- oder Radiomik-Pipeline aufbauen, können Sie nicht jedes Bild als austauschbar behandeln. Metadaten werden zu einer integralen Kontrollvariable: Zu den Eingabemerkmalen gehören häufig Modalität, kVp (Kilospannungsspitze), Hersteller, Kernel, Schichtdicke und sogar das Datum oder das Krankenhaus können von Bedeutung sein (Domänenverschiebung). Diese Metadatenfelder helfen dabei, Verzerrungen zu vermeiden, Daten zu harmonisieren und Bilder mit Kontext zu versehen. Viele Forscher bezeichnen Metadaten als „genauso wichtig wie Pixeldaten“.
Big Data bedeutet Skalierung. Das setzt unterschiedliche Quellen, mehrere Anbieter, unterschiedliche Institutionen und heterogene Formate voraus. DICOM-Metadaten sind die standardisierte „Sprache“, die dabei hilft, die Metadatenebene zu vereinheitlichen, die Suche/Indizierung zu ermöglichen, die Interoperabilität sicherzustellen und skalierbare Architekturen (Cloud-PACs, Verbundarchive) aufzubauen. Aber die Umsetzung ist wichtig. Dieselbe PMC-Studie ergab, dass viele Systeme den Standard nicht vollständig nutzen.
PostDicom bietet ein cloudbasiertes PACS (Picture Archiving and Communication System) zum Speichern, Anzeigen und Teilen von bildgebenden Studien und klinischen Dokumenten. Einige wichtige metadatenrelevante Funktionen von PostDicom:
• Unterstützung für DICOM-Tags und -Beschreibungen: Unsere Ressourcenbibliothek listet „DICOM-Modalität und -Tags“ auf, sodass Benutzer auf Tag-Listen und Beschreibungen zugreifen können. DICOM-Modalität und -Tags
• Api/Fhir-Integration: Es unterstützt API- und FHIR-Schnittstellen (Fast Healthcare Interoperability Resources), sodass Metadaten programmgesteuert abgefragt, in andere Systeme integriert und analysiert werden können.
• Cloud-Skalierbarkeit und gemeinsame Nutzung an mehreren Standorten: Gemeinsame Nutzung durch Patienten, Ärzte und Einrichtungen; dank unbegrenzter Skalierbarkeit werden Big-Data-Pipelines realisierbar.
• Erweiterte Bildverarbeitung und Multimodalitätsunterstützung: Dies betrifft zwar Pixel, aber die Unterstützung von Modalitäten wie PET-CT und Multi-Serien bedeutet, dass die Metadaten umfangreich sind (SUV-Werte, Fusionsvolumen, Modalitätstyp) und für Analysen verfügbar sind.
Mit einer Plattform wie PostDicom können Sie Metadaten über strukturierte Workflows, APIs und Cloud-Architekturen nutzen.
So strukturieren Sie den Arbeitsablauf von Roharchiven bis hin zu analysebereiten Erkenntnissen.
 - Created by PostDICOM.jpg)
Der erste Schritt bei der Nutzung von DICOM-Metadaten für Analysen ist die Extraktion und Normalisierung. Bibliotheken wie das Open-Source-Python-Paket PyDICOM werden häufig verwendet, um DICOM-Dateien zu analysieren und relevante Tags zu extrahieren, darunter Bildzeilen und -spalten, Faltungskerne und modalitätsspezifische Erfassungsparameter.
Der Umgang mit Heterogenität ist von entscheidender Bedeutung, da verschiedene Anbieter häufig private Tags oder nicht standardmäßige Implementierungen verwenden. Robustes Parsen, Fallback-Logik und umfassende Tag-Mapping-Tabellen sind erforderlich, um die Konsistenz aller Datensätze sicherzustellen.
Nach der Extraktion müssen die Metadaten normalisiert und Standardontologien und -strukturen wie Modalitätscodes, Anbieternamen, Schichtdickenkategorien und standardisierten Datums- und Uhrzeitformaten zugeordnet werden.
Schließlich sollten die strukturierten Metadaten in einer Big-Data-Umgebung gespeichert werden, z. B. in einer relationalen Datenbank, einem NoSQL-Speicher oder einem spaltenförmigen Data Lake, mit Indizierung, um schnelle und effiziente Abfragen zu ermöglichen.
Nach der Extraktion müssen Metadaten einer Qualitätssicherung unterzogen werden, um Genauigkeit und Zuverlässigkeit zu gewährleisten. Fehlende oder inkonsistente Felder, z. B. leere Schichtdickenwerte, inkonsistente Modalitätsbezeichnungen oder doppelte UIDs für Studieninstanzen, müssen identifiziert und korrigiert werden.
Datenschutz und Anonymisierung sind in dieser Phase ebenfalls von entscheidender Bedeutung, da Metadaten häufig personenbezogene Daten wie Patientennamen, IDs und Daten enthalten; Tools und Protokolle zur Anonymisierung sind unerlässlich.
Die Pflege umfassender Audit-Trails ist eine weitere wichtige Praxis. Sie dokumentiert, wann Metadaten extrahiert wurden, welche Parserversionen verwendet wurden und welche Fehler während des Prozesses aufgetreten sind.
Die Unternehmensrichtlinien sollten auch Pflichtfelder definieren und Leitlinien für den Umgang mit veralteten oder unvollständigen Datensätzen enthalten, um sicherzustellen, dass die nachgelagerten Analysen korrekt und gesetzeskonform sind.
Der nächste Schritt ist die metadatengestützte Indexierung und das Feature Engineering, bei dem Rohmetadaten in verwertbare Informationen umgewandelt werden.
Dazu gehören die Erstellung von Indizes und Filtern, die es Forschern und Analysten ermöglichen, bestimmte Datensätze abzufragen, z. B. alle Brust-CT-Scans mit einer Schichtdicke unter 1,5 Millimetern von einem bestimmten Anbieter innerhalb eines bestimmten Datumsbereichs abzurufen.
Das Feature Engineering baut darauf auf, indem es Metadatenfelder wie Hersteller, Modell, Erfassungsdatum, Schichtdicke, Faltungskern, Kontrastprotokoll, Körperregion, Strahlendosis und Institutions-ID zu strukturierten Variablen kombiniert, die für die Analyse geeignet sind.
Metadaten können auch mit klinischen Datensätzen verknüpft werden, wodurch Bildgebungsdaten mit Patientenergebnissen, Diagnosen oder Behandlungen verknüpft werden. Diese Verknüpfung ermöglicht eine ganzheitlichere Sicht auf Bildgebungsdaten und ihren klinischen Kontext.
Sobald die Metadaten indexiert und die Funktionen entwickelt sind, werden Analysen und die Generierung von Erkenntnissen möglich.
Deskriptive Analysen können das Studienvolumen nach Modalität, Anbieter oder Region aufdecken, Trends bei den Erfassungsparametern verfolgen und Fehler oder Inkonsistenzen bei bildgebenden Verfahren aufzeigen. Vergleichende Analysen ermöglichen die Bewertung von Erfassungsprotokollen in verschiedenen Einrichtungen, die Erkennung von Abweichungen und die Identifizierung von Ausreißer-Scans, die möglicherweise besondere Aufmerksamkeit erfordern.
Für maschinelles Lernen und KI-Anwendungen sind Metadaten unerlässlich, um den Domänenwechsel zu kontrollieren, sicherzustellen, dass Trainings- und Testdatensätze angemessen geschichtet sind, und um pixelbasierte Funktionen mit strukturierten Metadatenvariablen zu kombinieren. Operative Dashboards können diese Daten dann nutzen, um die Arbeitslast zu überwachen, Qualitätssicherungskennzahlen zu bewerten und die Einhaltung der Protokolle standortübergreifend sicherzustellen.
Schließlich vervollständigen Feedback und kontinuierliche Verbesserungen den Metadaten-Lebenszyklus. Aus Analysen gewonnene Erkenntnisse können in die Verfeinerung der Erfassungsprotokolle und die Standardisierung von Arbeitsabläufen einfließen, um die allgemeine Datenqualität zu verbessern.
Neue Bildgebungsstudien und Metadaten sollten kontinuierlich aufgenommen werden, wobei die Leistung, die Abfragezeiten und die Datenintegrität des Metadatenspeichers überwacht werden sollten. Die gewonnenen Erkenntnisse sollten archiviert werden, um prädiktive Metadatenfelder zu erfassen, wiederkehrende Lücken oder Fehler zu beheben und die Governance-Praktiken zu verbessern.
Dieser iterative Ansatz stellt sicher, dass Metadaten-Pipelines robust, skalierbar und wertvoll für zukünftige Forschungen, KI-Anwendungen und betriebliche Entscheidungen bleiben.
• Variabilität zwischen Anbietern und Institutionen: Private Tags oder lose Standardinterpretationen.
• Fehlende oder beschädigte Metadaten: Ältere Studien haben möglicherweise unvollständige Header.
• Datenschutz und Anonymisierung: PHI müssen für Recherchen an mehreren Standorten anonymisiert werden.
• Skalierung und Leistung: Millionen von Bildern erfordern eine effiziente Verarbeitung und Speicherung.
• Domain Shift/Bias: Dominante Anbieter/Protokolle können KI-Modelle verzerren.
• Regulatorische und Compliance-Probleme: Bereitstellungen in mehreren Regionen können HIPAA-, GDPR- oder lokale Vorschriften beinhalten.
DICOM-Metadaten sind das verborgene Gerüst der Bildgebungsanalytik. Plattformen wie PostDICOM veranschaulichen, wie ein fragmentiertes Archiv von DICOM-Dateien in ein durchsuchbares, skalierbares, metadatengestütztes Ökosystem umgewandelt werden kann. Wenn Sie PostDicom erkunden möchten, empfehlen wir Ihnen, unsere kostenlose 7-Tage-Testversion zu nutzen.