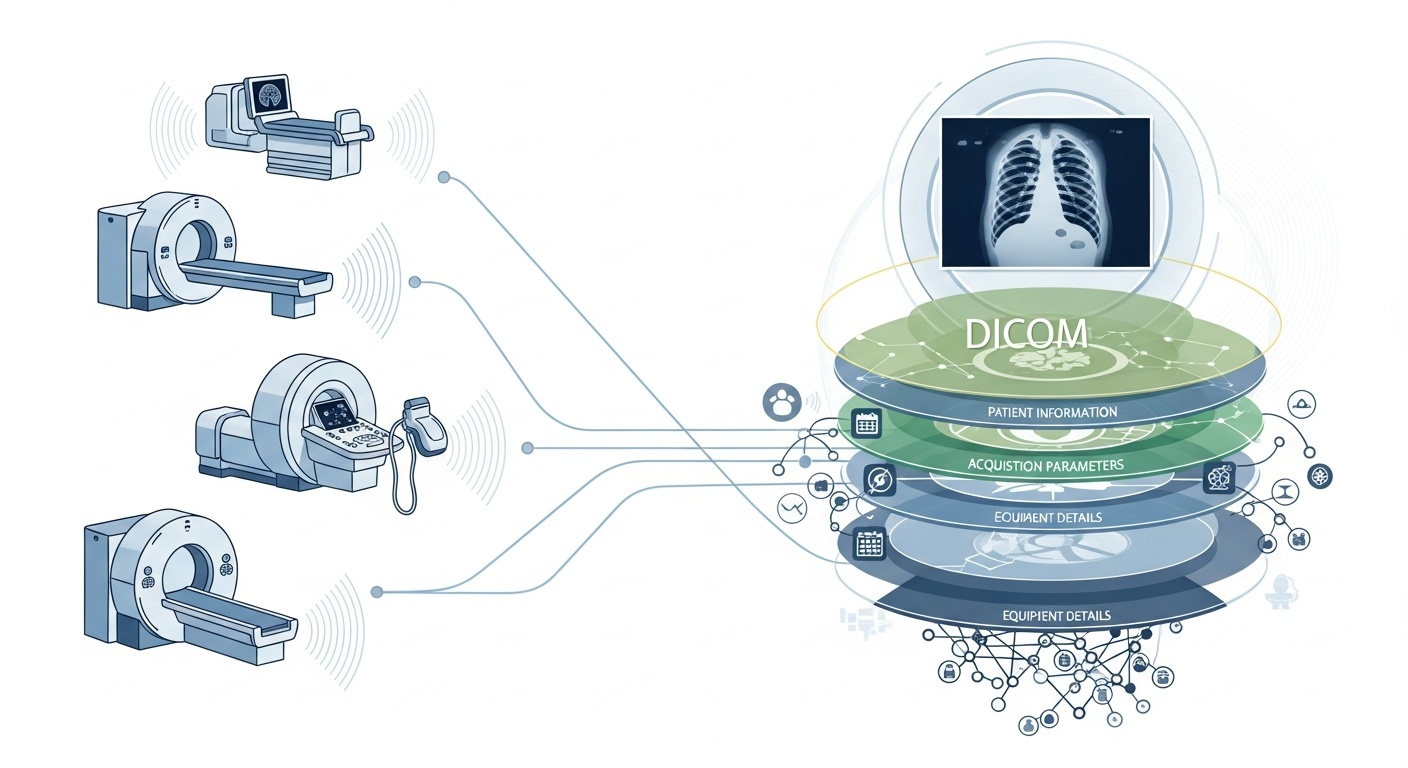
Za każdym razem, gdy zespół radiologiczny bada pacjenta, na przykład wykonując TK, rezonans magnetyczny lub USG, generowana jest kaskada danych. Najbardziej widoczny jest sam obraz, ale za nim kryje się bogata warstwa metadanych: kto, co, kiedy, jak i gdzie wykonał badanie. Warstwą tą zarządza standard DICOM (Digital Imaging and Communications in Medicine), standard formatu obrazowania medycznego opracowany dekady temu przez National Electrical Manufacturers Association (NEMA) i American College of Radiology (ACR).
To, co czyni metadane tak interesującymi, to fakt, że są one ustrukturyzowane, czytelne maszynowo i niezwykle szczegółowe: ustawienia sprzętu, parametry akwizycji, dane demograficzne pacjenta, identyfikatory badań, a nawet kody placówek i szczegóły dotyczące producenta modalności. To bogactwo umożliwia analitykę Big Data, badania, modelowanie AI i standaryzację protokołów, jeśli zostanie dobrze wykorzystane.
W kontekście Big Data nie mówimy tylko o kilkudziesięciu badaniach obrazowych. Mówimy o setkach tysięcy, a nawet milionach obrazów z różnych modalności, lokalizacji i od różnych dostawców, przy czym metadane stanowią kluczową warstwę indeksującą: „kto, kiedy i jak” dla każdego elementu. Bez skutecznego wykorzystania metadanych, ryzykują Państwo posiadanie ogromnego archiwum obrazów z minimalną możliwością ich przeszukiwania, porównywania lub wyciągania z nich wniosków. Niedawny przegląd stwierdza: „większość informacji przechowywanych w archiwach PACS nigdy nie jest ponownie otwierana”, co jest zmarnowaną szansą.
Załóżmy, że prowadzą Państwo wieloośrodkowe badanie tomografii komputerowej płuc w celu wczesnego wykrywania rozedmy. Będą Państwo chcieli wybrać skany na podstawie parametrów: sprzedawca/marka/model skanera, grubość warstwy, jądro rekonstrukcji, wiek pacjenta, zakresy dat, a może nawet parametry dawki. Większość z nich to pola metadanych, a nie dane pikselowe. Ekstrakcja tych tagów umożliwia zbudowanie kohorty, wykluczenie niekompatybilnych skanów (np. zbyt grubych warstw) i zapewnienie porównywalności.
Metadane pozwalają monitorować sam proces obrazowania: czy placówka stosuje właściwy protokół? Czy parametry akwizycji zmieniają się w czasie (np. pole widzenia, czas podania kontrastu)? Czy ustawienia dostawcy są spójne? W świecie Big Data, gdzie codziennie wykonuje się tysiące skanów, nie można polegać na ludzkiej ocenie wzrokowej. Potrzebna jest analityka metadanych. Wiele systemów PACS niedostatecznie wykorzystuje metadane z tego powodu.
Jeśli budują Państwo potok AI lub radiomiki, nie mogą Państwo traktować każdego obrazu jako zamiennego. Metadane stają się integralną zmienną kontrolną: cechy wejściowe często obejmują modalność, kVp (szczytowe kilowolty), producenta, jądro, grubość warstwy, a nawet data lub szpital mogą mieć znaczenie (przesunięcie domeny). Te pola metadanych pomagają zarządzać stronniczością, harmonizować dane i dodawać adnotacje z kontekstem do obrazów. Wielu badaczy nazywa metadane „równie ważnymi jak dane pikselowe.”
Big Data oznacza skalę. Wiąże się to z różnymi źródłami, wieloma dostawcami, różnymi instytucjami i heterogenicznymi formatami. Metadane DICOM to ustandaryzowany „język”, który pomaga ujednolicić warstwę metadanych, umożliwia wyszukiwanie/indeksowanie, zapewnia interoperacyjność i budowanie skalowalnych architektur (Cloud PACS, sfederowane archiwa). Ale wdrożenie ma znaczenie. To samo badanie PMC wykazało, że wiele systemów nie w pełni wykorzystuje ten standard.
PostDICOM oferuje oparty na chmurze system PACS (System Archiwizacji i Transmisji Obrazów) do przechowywania, przeglądania i udostępniania badań obrazowych oraz dokumentów klinicznych. Oto niektóre kluczowe funkcje PostDICOM związane z metadanymi:
• Obsługa tagów i opisów DICOM: Nasza biblioteka zasobów zawiera „Modalność i tagi DICOM”, umożliwiając użytkownikom dostęp do list tagów i opisów.Modalność i tagi DICOM
• Integracja API / FHIR: Obsługuje interfejsy API i FHIR (Fast Healthcare Interoperability Resources), umożliwiając programowe odpytywanie metadanych, integrację z innymi systemami i analizę.
• Skalowalność w chmurze i udostępnianie między placówkami: Udostępnianie pacjentom, lekarzom i instytucjom; nieograniczona skalowalność oznacza, że potoki Big Data stają się wykonalne.
• Zaawansowane przetwarzanie obrazu i obsługa wielu modalności: Chociaż dotyczy to pikseli, obsługa modalności takich jak PET–CT i serii wielokrotnych oznacza, że metadane są istotne (wartości SUV, objętości fuzji, typ modalności) i dostępne do analizy.
Korzystanie z platformy takiej jak PostDICOM pozwala Państwu wykorzystać metadane poprzez ustrukturyzowane przepływy pracy, interfejsy API i architekturę chmury.
Oto jak ustrukturyzować przepływ pracy od surowych archiwów do wniosków gotowych do analizy.
 - Created by PostDICOM.jpg)
Pierwszym krokiem w wykorzystaniu metadanych DICOM do analityki jest ekstrakcja i normalizacja. Biblioteki takie jak open-source'owy pakiet Python PyDicom są powszechnie używane do parsowania plików DICOM i wyodrębniania odpowiednich tagów, w tym rzędów i kolumn obrazu, jąder splotu i parametrów akwizycji specyficznych dla danej modalności.
Obsługa heterogeniczności jest kluczowa, ponieważ różni dostawcy często używają tagów prywatnych lub niestandardowych implementacji. Solidne parsowanie, logika awaryjna i kompleksowe tabele mapowania tagów są wymagane, aby zapewnić spójność w zestawach danych.
Po wyodrębnieniu metadane muszą zostać znormalizowane i zmapowane do standardowych ontologii i struktur, takich jak kody modalności, nazwy dostawców, kategorie grubości warstwy oraz ustandaryzowane formaty daty i czasu.
Ostatecznie ustrukturyzowane metadane powinny być przechowywane w środowisku Big Data, takim jak relacyjna baza danych, magazyn NoSQL lub jezioro danych kolumnowych, z indeksowaniem umożliwiającym szybkie i wydajne wyszukiwanie.
Po wyodrębnieniu metadane muszą przejść kontrolę jakości, aby zapewnić dokładność i niezawodność. Brakujące lub niespójne pola, takie jak puste wartości grubości warstwy, niespójne etykiety modalności lub zduplikowane UID instancji badania, muszą zostać zidentyfikowane i poprawione.
Prywatność i anonimizacja są również krytyczne na tym etapie, ponieważ metadane często zawierają dane osobowe, w tym nazwiska pacjentów, identyfikatory i daty; narzędzia i protokoły deidentyfikacji są niezbędne.
Utrzymywanie kompleksowych ścieżek audytu to kolejna ważna praktyka, dokumentująca, kiedy metadane zostały wyodrębnione, jakie wersje parserów zostały użyte i wszelkie błędy napotkane podczas procesu.
Polityki zarządzania powinny również definiować pola obowiązkowe i dostarczać wskazówek, jak postępować ze starszymi lub niekompletnymi zestawami danych, aby zapewnić, że dalsza analityka będzie dokładna i zgodna z przepisami.
Następnym krokiem jest indeksowanie oparte na metadanych i inżynieria cech, która przekształca surowe metadane w użyteczne informacje.
Obejmuje to tworzenie indeksów i filtrów, które pozwalają badaczom i analitykom wyszukiwać określone zestawy danych, na przykład pobierać wszystkie skany TK klatki piersiowej o grubości warstwy poniżej 1,5 milimetra od konkretnego dostawcy w określonym zakresie dat.
Inżynieria cech opiera się na tym, łącząc pola metadanych, takie jak dostawca, model, data akwizycji, grubość warstwy, jądro splotu, protokół kontrastu, obszar ciała, dawka promieniowania i identyfikator instytucji, w ustrukturyzowane zmienne odpowiednie do analizy.
Metadane można również łączyć z klinicznymi zestawami danych, powiązując dane obrazowe z wynikami pacjentów, diagnozami lub leczeniem. To powiązanie pozwala na bardziej całościowe spojrzenie na dane obrazowe i ich kontekst kliniczny.
Po zindeksowaniu metadanych i zaprojektowaniu cech możliwa staje się analityka i generowanie wniosków.
Analityka opisowa może ujawnić wolumeny badań według modalności, dostawcy lub regionu, śledzić trendy w parametrach akwizycji i podkreślać błędy lub niespójności w praktykach obrazowania. Analityka porównawcza umożliwia ocenę protokołów akwizycji w różnych instytucjach, wykrywanie odchyleń i identyfikację skanów odstających, które mogą wymagać szczególnej uwagi.
W przypadku zastosowań uczenia maszynowego i AI, metadane są niezbędne do kontrolowania przesunięcia domeny, zapewnienia, że zestawy danych treningowych i testowych są odpowiednio stratyfikowane, oraz łączenia cech opartych na pikselach ze zmiennymi strukturalnymi metadanych. Pulpity operacyjne mogą następnie wykorzystywać te dane do monitorowania obciążenia pracą, oceny metryk zapewnienia jakości i zapewnienia zgodności z protokołami w różnych lokalizacjach.
Wreszcie, informacje zwrotne i ciągłe doskonalenie zamykają cykl życia metadanych. Wnioski płynące z analityki mogą informować o udoskonalaniu protokołów akwizycji i standaryzacji przepływów pracy w celu poprawy ogólnej jakości danych.
Nowe badania obrazowe i metadane powinny być stale przyjmowane, przy monitorowaniu wydajności magazynu metadanych, czasów zapytań i integralności danych. Wyciągnięte wnioski powinny być archiwizowane w celu przechwycenia predykcyjnych pól metadanych, rozwiązania powtarzających się luk lub błędów i poprawy praktyk zarządzania.
To iteracyjne podejście zapewnia, że potoki metadanych pozostają solidne, skalowalne i wartościowe dla przyszłych badań, zastosowań AI i podejmowania decyzji operacyjnych.
• Zmienność dostawców/instytucji: Tagi prywatne lub luźne interpretacje standardów.
• Brakujące lub uszkodzone metadane: Starsze badania mogą mieć niekompletne nagłówki.
• Prywatność danych i anonimizacja: PHI muszą zostać zanonimizowane dla badań wieloośrodkowych.
• Skala i wydajność: Miliony obrazów wymagają wydajnego przetwarzania i przechowywania.
• Przesunięcie domeny/stronniczość: Dominujący dostawcy/protokoły mogą zniekształcać modele AI.
• Kwestie regulacyjne i zgodności: Wdrożenia w wielu regionach mogą wiązać się z HIPAA, RODO lub lokalnymi przepisami.
Metadane DICOM to ukryty szkielet analityki obrazowania. Platformy takie jak PostDICOM pokazują, jak przekształcić sfragmentowane archiwum plików DICOM w przeszukiwalny, skalowalny ekosystem oparty na metadanych. Jeśli chcą Państwo poznać PostDICOM, zachęcamy do skorzystania z naszego 7-dniowego bezpłatnego okresu próbnego.