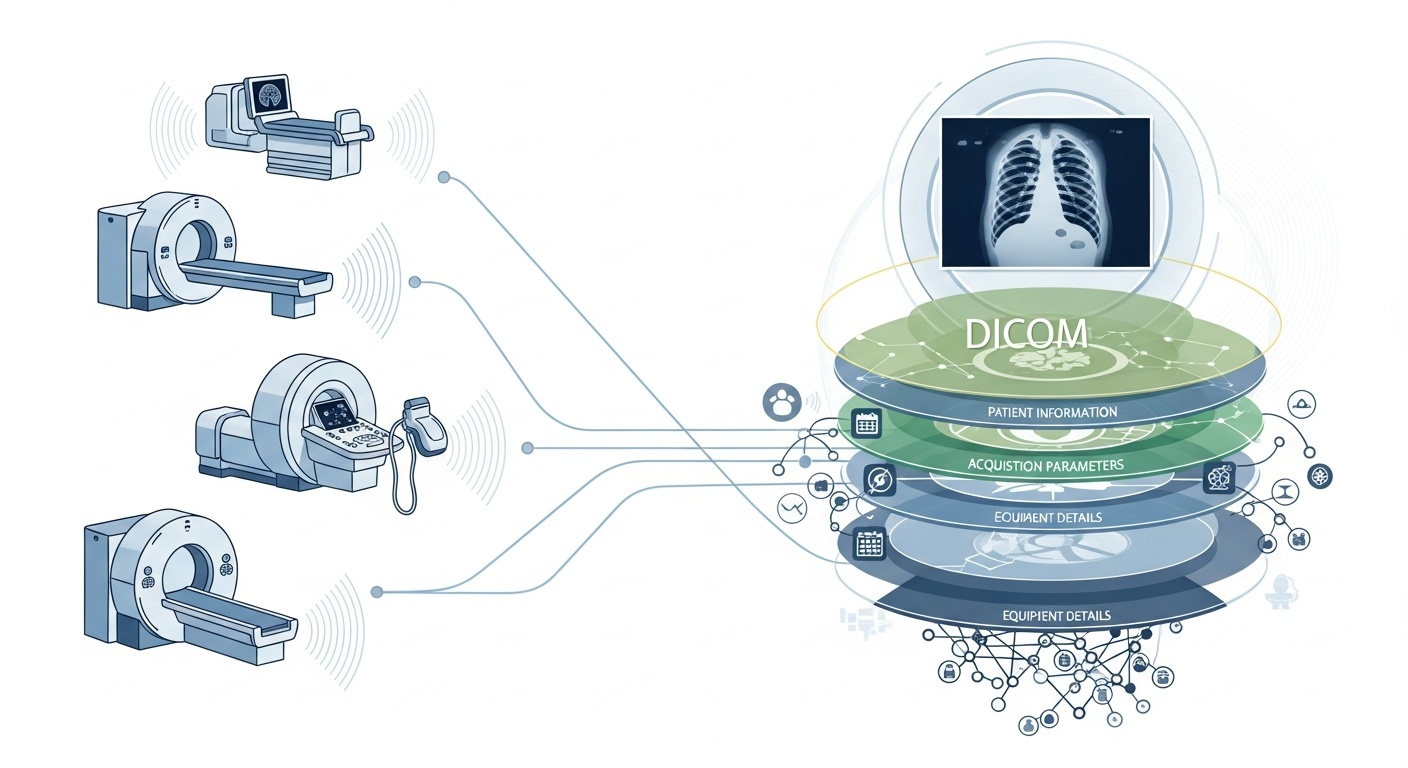
Hver gang et radiologiteam skanner en pasient, for eksempel CT, MR eller ultralyd, genereres en kaskade av data. Mest synlig er selve bildet, men bak det ligger et rikt lag av metadata: hvem, hva, når, hvordan og hvor for skanningen. Dette laget styres av standarden DICOM (Digital Imaging and Communications in Medicine), en standard for medisinsk bildeformat utarbeidet av National Electrical Manufacturers Association (NEMA) og American College of Radiology (ACR) for flere tiår siden.
Det som gjør metadataene så interessante, er at de er strukturerte, maskinlesbare og bemerkelsesverdig detaljerte: utstyrsinnstillinger, opptaksparametere, pasientdemografi, studie-ID-er, til og med institusjonskoder og detaljer om modalitetsprodusenten. Denne rikdommen er det som muliggjør big data-analyse, nedstrømsforskning, AI-modellering og protokollstandardisering hvis du utnytter det godt.
I sammenheng med big data snakker vi ikke bare om noen titalls bildeundersøkelser. Vi snakker om hundretusenvis, eller til og med millioner, av bilder på tvers av modaliteter, steder og leverandører, med metadata som det viktigste indekseringslaget: "hvem & når & hvordan" for hvert element. Uten effektiv bruk av metadata risikerer du å ha et massivt arkiv med bilder, men minimal evne til å søke, sammenligne eller hente innsikt fra dem. En nylig gjennomgang sier: "majoriteten av informasjonen lagret i PACS-arkiver blir aldri åpnet igjen," en bortkastet mulighet.
Si at du kjører en studie på tvers av flere steder med lunge-CT-skanninger for tidlig påvisning av emfysem. Du vil ønske å velge skanninger basert på parametere: skannerleverandør/merke/modell, snitykkelse, rekonstruksjonskjerne, pasientalder, datointervaller, kanskje til og med doseparametere. De fleste av disse er metadatafelt, ikke pikseldata. Å trekke ut disse taggene gjør det mulig å bygge kohorten, ekskludere inkompatible skanninger (f.eks. for tykke snitt) og sikre sammenlignbarhet.
Metadata lar deg overvåke selve bildeprosessen: bruker institusjonen riktig protokoll? Avviker opptaksparametere over tid (f.eks. synsfelt, timing av kontrastinjeksjon)? Er leverandørinnstillingene konsekvente? I en big data-verden der tusenvis av skanninger skjer daglig, kan du ikke stole på menneskelig øyemål. Du trenger analyse av metadata. Mange PACS-systemer underutnytter metadata av denne grunnen.
Hvis du bygger en AI- eller radiomikk-pipeline, kan du ikke behandle hvert bilde som utskiftbart. Metadata blir en integrert kontrollvariabel: inndatafunksjoner inkluderer ofte modalitet, kVp (kilovoltage peak), produsent, kjerne, snitykkelse, og til og med dato eller sykehus kan ha betydning (domeneskifte). Disse metadatafeltene bidrar til å håndtere skjevhet, harmonisere data og annotere bilder med kontekst. Mange forskere kaller metadata "like viktig som pikseldata."
Big data betyr skala. Det innebærer varierte kilder, flere leverandører, forskjellige institusjoner og heterogene formater. DICOM-metadata er det standardiserte "språket" som bidrar til å forene metadatalaget, muliggjøre søk/indeksering, sikre interoperabilitet og bygge skalerbare arkitekturer (Cloud PACS, fødererte arkiver). Men implementering er viktig. Den samme PMC-studien fant at mange systemer ikke utnytter standarden fullt ut.
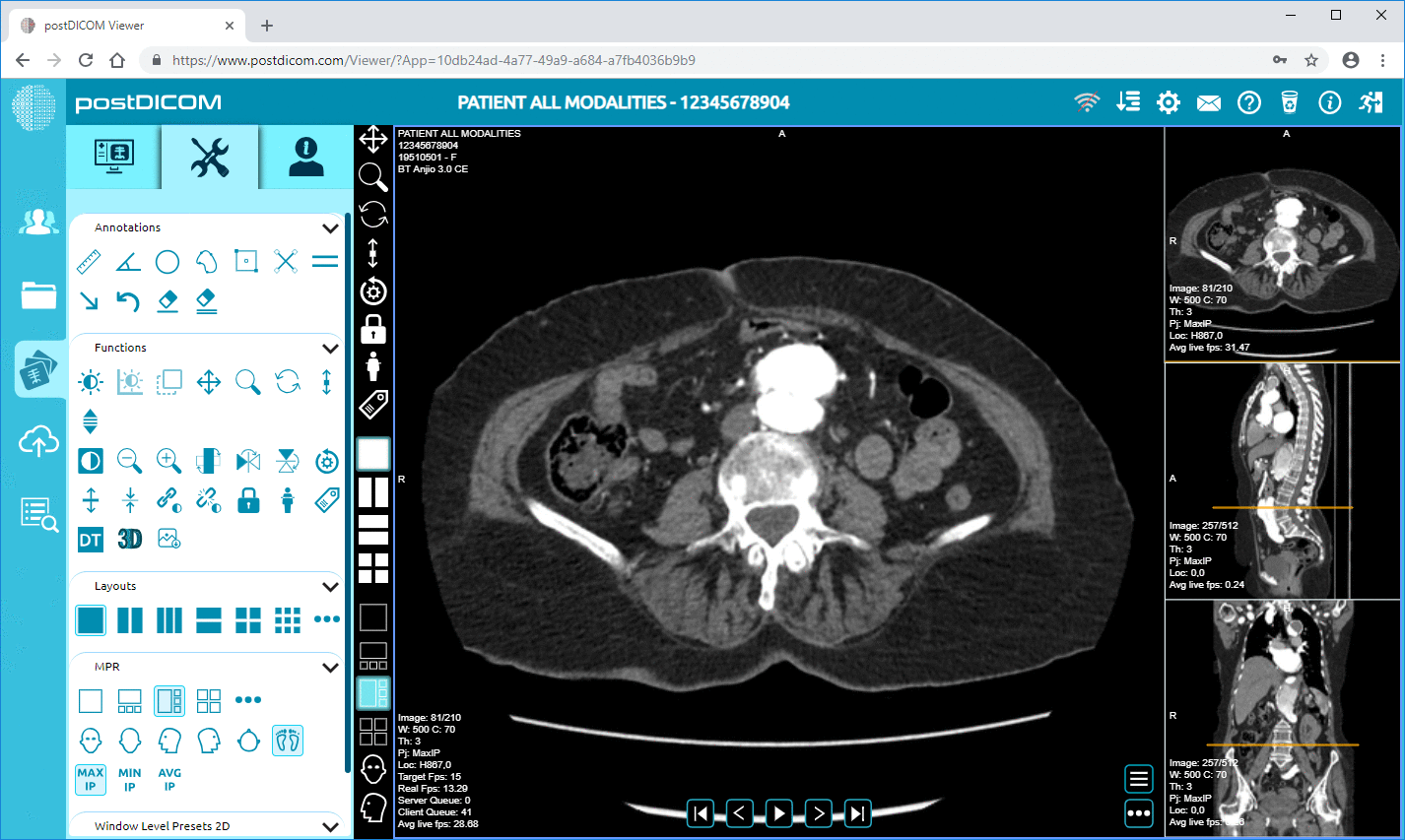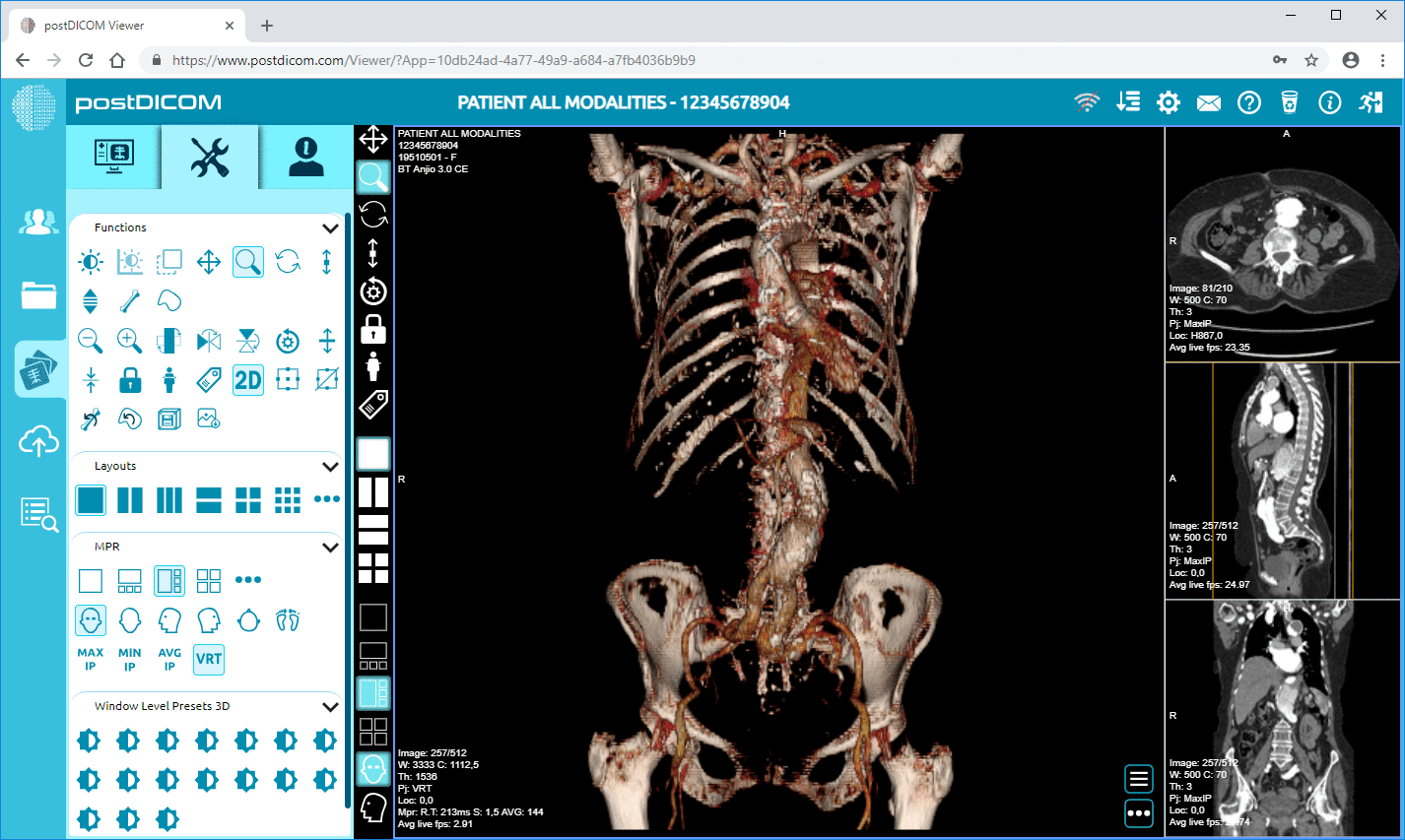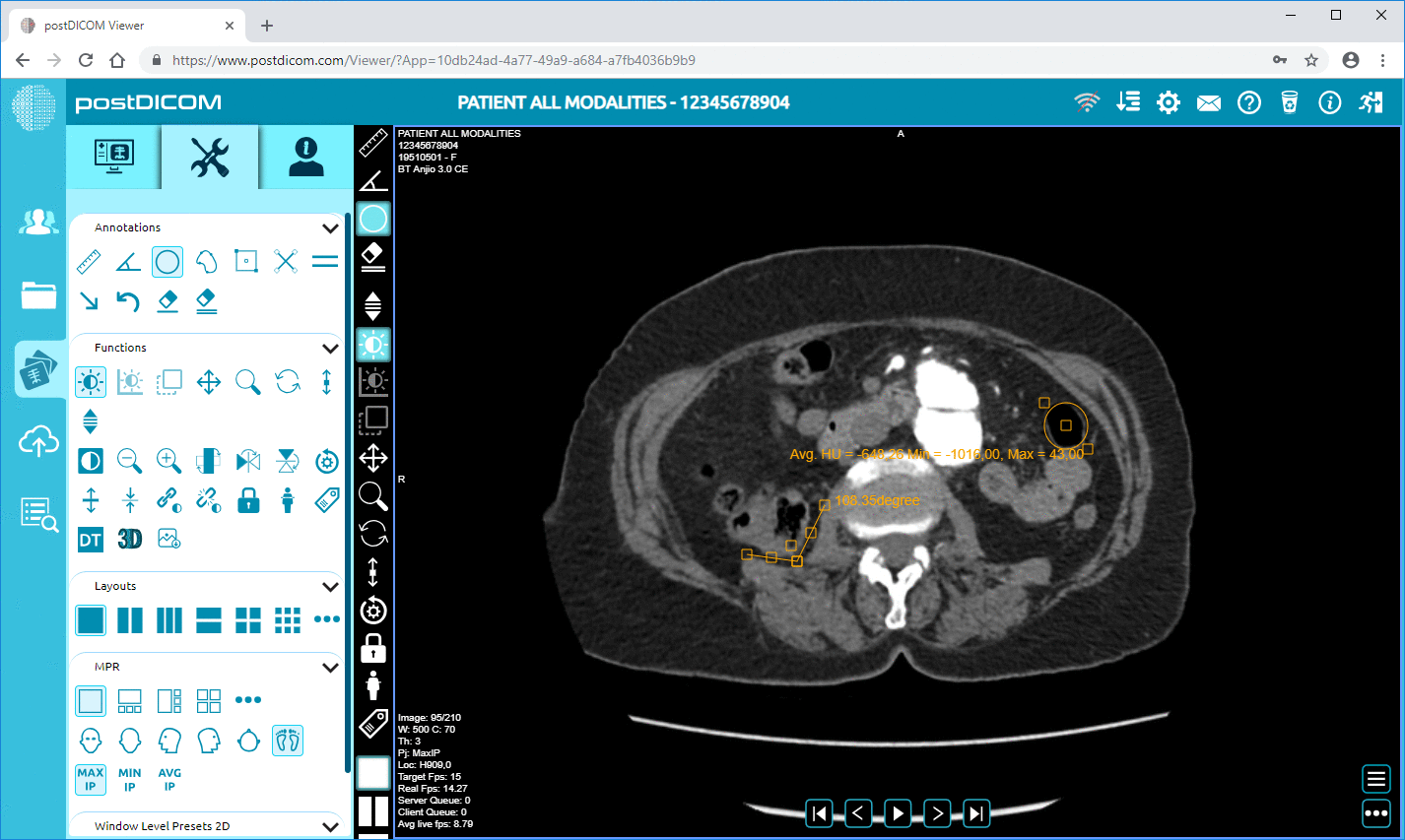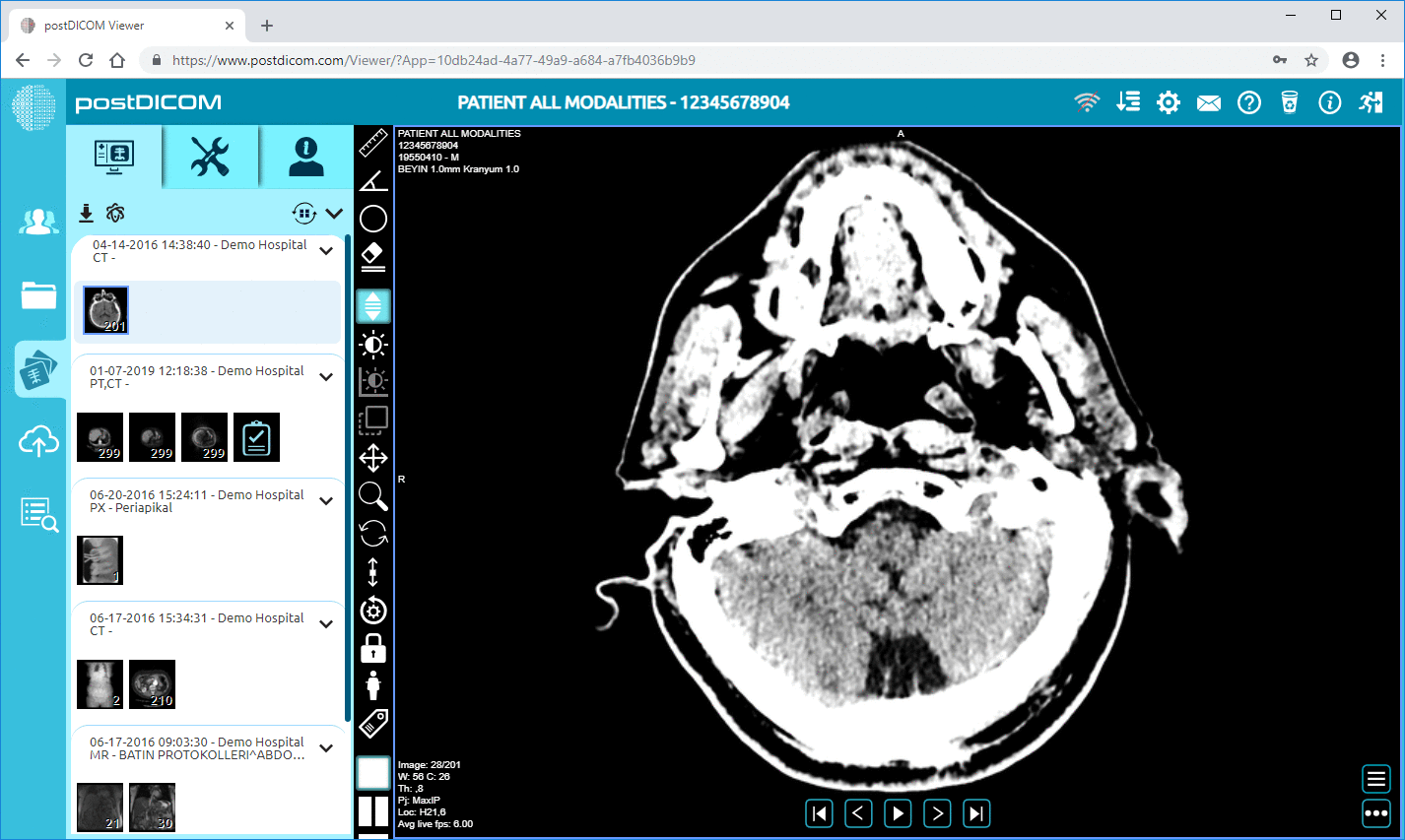
PostDICOM tilbyr en skybasert PACS (Picture Archiving and Communication System) for lagring, visning og deling av bildeundersøkelser og kliniske dokumenter. Noen viktige metadata-relevante funksjoner i PostDICOM:
• Støtte for DICOM-tagger og beskrivelser: Vårt ressursbibliotek viser "DICOM-modalitet og tagger", som lar brukere få tilgang til tagglister og beskrivelser.DICOM-modalitet og tagger
• API / FHIR-integrasjon: Den støtter API- og FHIR-grensesnitt (Fast Healthcare Interoperability Resources), som gjør at metadata kan programmeres for forespørsel, integreres med andre systemer og analyseres.
• Skalerbarhet i skyen og deling på tvers av steder: Deling på tvers av pasienter, leger, institusjoner; ubegrenset skalerbarhet betyr at big data-pipelines blir gjennomførbare.
• Avansert bildebehandling og multimodalitetsstøtte: Selv om dette gjelder piksler, betyr støtten for modaliteter som PET–CT og multiserier at metadataene er omfattende (SUV-verdier, fusjonsvolumer, modalitetstype) og tilgjengelige for analyse.
Bruk av en plattform som PostDICOM lar deg utnytte metadata gjennom strukturerte arbeidsflyter, API-er og skyarkitektur.
Her er hvordan du strukturerer arbeidsflyten fra råarkiver til analyseklare innsikter.
 - Created by PostDICOM.jpg)
Det første trinnet i å utnytte DICOM-metadata for analyse er utvinning og normalisering. Biblioteker som den åpen kildekode-baserte Python-pakken PyDicom brukes ofte til å parse DICOM-filer og trekke ut relevante tagger, inkludert bilderader og kolonner, konvolusjonskjerner og modalitetsspesifikke opptaksparametere.
Håndtering av heterogenitet er avgjørende, ettersom forskjellige leverandører ofte bruker private tagger eller ikke-standard implementeringer. Robust parsing, fallback-logikk og omfattende tagg-kartleggingstabeller kreves for å sikre konsistens på tvers av datasett.
Når metadataene er trukket ut, må de normaliseres og kartlegges til standard ontologier og strukturer, som modalitetskoder, leverandørnavn, kategorier for snitykkelse og standardiserte dato- og tidsformater.
Til slutt bør de strukturerte metadataene lagres i et big data-miljø, som en relasjonsdatabase, NoSQL-lager eller kolonnebasert datasjø, med indeksering for å muliggjøre raske og effektive forespørsler.
Når metadataene er trukket ut, må de gjennomgå kvalitetssikring for å sikre nøyaktighet og pålitelighet. Manglende eller inkonsekvente felt, som blanke verdier for snitykkelse, inkonsekvente modalitetsetiketter eller dupliserte Study Instance UIDs, må identifiseres og korrigeres.
Personvern og anonymisering er også kritisk i denne fasen, da metadata ofte inneholder personlig identifiserbar informasjon inkludert pasientnavn, ID-er og datoer; avidentifiseringsverktøy og protokoller er essensielle.
Å opprettholde omfattende revisjonsspor er en annen viktig praksis, som dokumenterer når metadata ble trukket ut, hvilke parserversjoner som ble brukt, og eventuelle feil som oppstod under prosessen.
Styringspolicyer bør også definere obligatoriske felt og gi veiledning om hvordan man skal håndtere eldre eller ufullstendige datasett for å sikre at nedstrømsanalyser er nøyaktige og i samsvar med regelverket.
Neste trinn er metadatadrevet indeksering og funksjonsutvikling, som forvandler rå metadata til handlingsrettet informasjon.
Dette innebærer å lage indekser og filtre som lar forskere og analytikere spørre etter spesifikke datasett, for eksempel å hente alle thorax-CT-skanninger med snitykkelse under 1,5 millimeter fra en bestemt leverandør innenfor et gitt datointervall.
Funksjonsutvikling bygger på dette ved å kombinere metadatafelt som leverandør, modell, opptaksdato, snitykkelse, konvolusjonskjerne, kontrastprotokoll, kroppsregion, strålingsdose og institusjons-ID til strukturerte variabler egnet for analyse.
Metadata kan også kobles til kliniske datasett, som kobler bildedata til pasientutfall, diagnoser eller behandlinger. Denne koblingen gir et mer helhetlig syn på bildedata og deres kliniske kontekst.
Når metadata er indeksert og funksjoner er utviklet, blir analyse og generering av innsikt mulig.
Beskrivende analyser kan avsløre studievolumer etter modalitet, leverandør eller region, spore trender i opptaksparametere og fremheve feil eller inkonsekvenser i bildepraksis. Komparative analyser muliggjør evaluering av opptaksprotokoller på tvers av institusjoner, deteksjon av avvik og identifisering av unormale skanninger som kan kreve spesiell oppmerksomhet.
For maskinlæring og AI-applikasjoner er metadata avgjørende for å kontrollere domeneskifte, sikre at trenings- og testdatasett stratifiseres riktig, og kombinere pikselbaserte funksjoner med strukturerte metadatavariabler. Operasjonelle dashbord kan deretter utnytte disse dataene til å overvåke arbeidsbelastning, vurdere kvalitetssikringsmålinger og sikre protokolleetterlevelse på tvers av steder.
Til slutt fullfører tilbakemelding og kontinuerlig forbedring metadatalivssyklusen. Innsikt fra analyser kan informere om forbedring av opptaksprotokoller og standardisering av arbeidsflyter for å forbedre den generelle datakvaliteten.
Nye bildeundersøkelser og metadata bør inntas kontinuerlig, med overvåking av metadatalagerets ytelse, responstider og dataintegritet. Lærdom bør arkiveres for å fange opp prediktive metadatafelt, adressere tilbakevendende hull eller feil, og forbedre styringspraksis.
Denne iterative tilnærmingen sikrer at metadatapipelines forblir robuste, skalerbare og verdifulle for fremtidig forskning, AI-applikasjoner og operasjonelle beslutninger.
• Leverandør-/institusjonsvariabilitet: Private tagger eller løse tolkninger av standarder.
• Manglende eller korrupte metadata: Eldre studier kan ha ufullstendige overskrifter.
• Personvern og anonymisering: Helseopplysninger (PHI) må avidentifiseres for forskning på tvers av steder.
• Skala og ytelse: Millioner av bilder krever effektiv behandling og lagring.
• Domeneskifte/skjevhet: Dominerende leverandører/protokoller kan skape skjevheter i AI-modeller.
• Regulatoriske og samsvarsmessige problemer: Utplassering i flere regioner kan involvere HIPAA, GDPR eller lokale forskrifter.
DICOM-metadata er det skjulte skjelettet i bildeanalyse. Plattformer som PostDICOM illustrerer hvordan man kan forvandle et fragmentert arkiv av DICOM-filer til et søkbart, skalerbart, metadatadrevet økosystem. Hvis du vil utforske PostDICOM, oppfordrer vi deg til å prøve vår 7-dagers gratis prøveperiode.
|
Cloud PACS og online DICOM-visningsprogramLast opp DICOM-bilder og medisinske dokumenter til PostDICOM-servere. Lagre, vis, samarbeid og del dine medisinske bildefiler. |