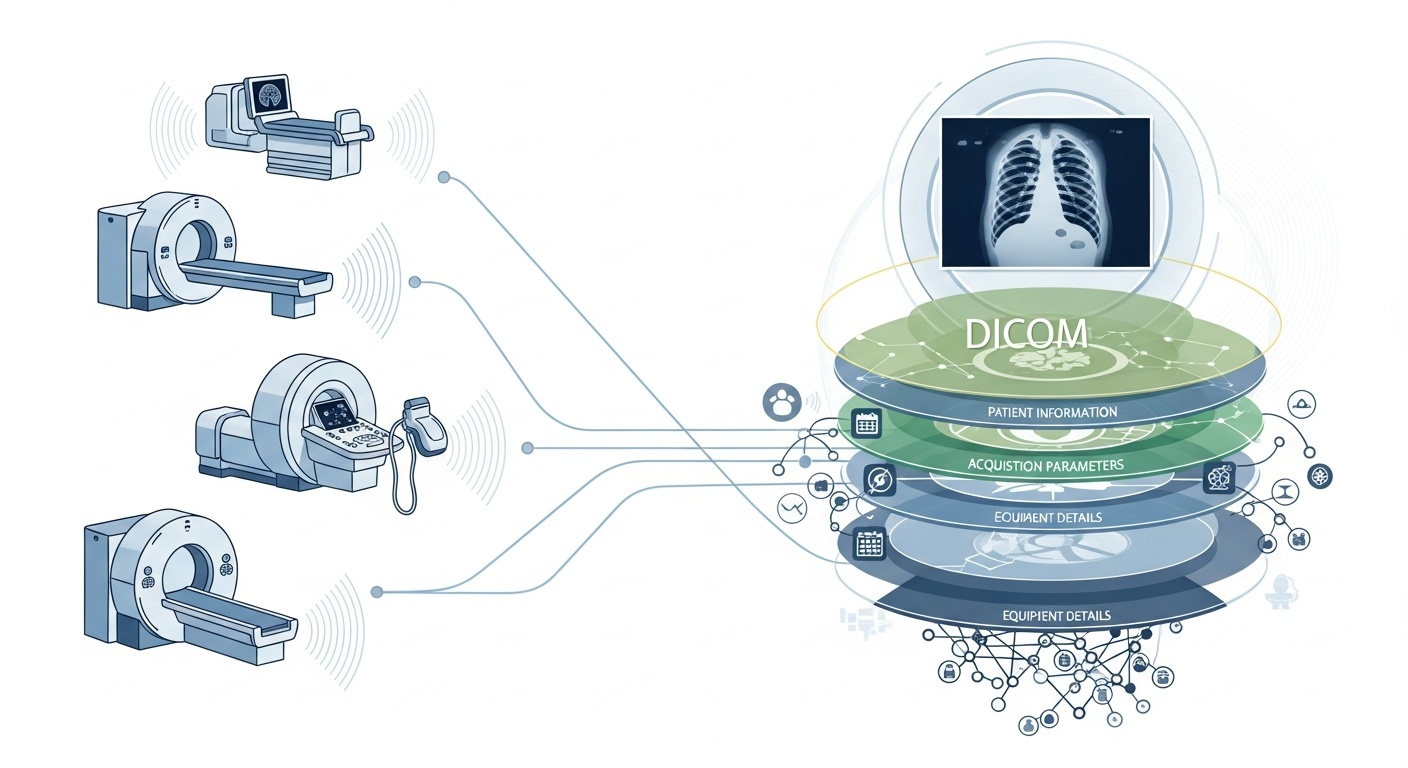
Chaque fois qu'une équipe de radiologie scanne un patient, par exemple un scanner, une IRM ou une échographie, une cascade de données est générée. L'image elle-même est la plus visible, mais derrière elle se trouve une riche couche de métadonnées : qui, quoi, quand, comment et où a été numérisée. Cette couche est régie par la norme DICOM (Digital Imaging and Communications in Medicine), une norme de format d'imagerie médicale mise au point par la National Electrical Manufacturers Association (NEMA) et l'American College of Radiology (ACR) il y a des décennies.
Ce qui rend les métadonnées si intéressantes, c'est qu'elles sont structurées, lisibles par machine et remarquablement détaillées : paramètres de l'équipement, paramètres d'acquisition, données démographiques des patients, numéros d'étude, et même codes d'établissement et détails du fabricant de la modalité. C'est cette richesse qui permet l'analyse des mégadonnées, la recherche en aval, la modélisation de l'IA et la standardisation des protocoles si vous l'exploitez bien.
Dans le contexte des mégadonnées, il ne s'agit pas simplement de quelques dizaines d'études d'imagerie. Nous parlons de centaines de milliers, voire de millions, d'images provenant de différents modes, sites et fournisseurs, avec des métadonnées comme couche d'indexation clé : « qui, quand et comment » de chaque article. Si vous n'utilisez pas efficacement les métadonnées, vous risquez de disposer d'une énorme archive d'images mais d'une capacité minimale à les interroger, à les comparer ou à en tirer des informations. Une étude récente indique que « la majorité des informations stockées dans les archives PACS ne sont plus jamais consultées », une occasion manquée.
Supposons que vous meniez une étude multisite sur les tomodensitogrammes pulmonaires pour la détection précoce de l'emphysème. Vous devrez sélectionner les scans en fonction de paramètres : fournisseur/marque/modèle du scanner, épaisseur de la tranche, noyau de reconstruction, âge du patient, plages de dates, peut-être même paramètres de dose. La plupart d'entre eux sont des champs de métadonnées et non des données en pixels. L'extraction de ces balises vous permet de créer la cohorte, d'exclure les scans incompatibles (par exemple, des tranches trop épaisses) et de garantir la comparabilité.
Les métadonnées vous permettent de surveiller le processus d'imagerie lui-même : l'établissement utilise-t-il le bon protocole ? Les paramètres d'acquisition varient-ils au fil du temps (par exemple, champ de vision, moment d'injection du produit de contraste) ? Les paramètres du fournisseur sont-ils cohérents ? Dans un monde de mégadonnées où des milliers de scans sont effectués chaque jour, vous ne pouvez pas vous fier à l'observation humaine. Vous avez besoin d'analyses sur les métadonnées. De nombreux systèmes PACS sous-exploitent les métadonnées pour cette raison.
Si vous créez un pipeline d'IA ou de radiomique, vous ne pouvez pas traiter toutes les images comme interchangeables. Les métadonnées deviennent une variable de contrôle intégrale : les caractéristiques d'entrée incluent souvent la modalité, le kVp (pic de kilotension), le fabricant, le grain, l'épaisseur des tranches, et même la date ou l'hôpital peuvent avoir de l'importance (changement de domaine). Ces champs de métadonnées permettent de gérer les biais, d'harmoniser les données et d'annoter les images en fonction du contexte. De nombreux chercheurs considèrent que les métadonnées sont « aussi importantes que les données en pixels ».
Les mégadonnées sont synonymes d'évolutivité. Cela implique des sources variées, de multiples fournisseurs, des institutions différentes et des formats hétérogènes. Les métadonnées DICOM sont le « langage » standardisé qui permet d'unifier la couche de métadonnées, de permettre la recherche/l'indexation, de garantir l'interopérabilité et de créer des architectures évolutives (Cloud-PACS, archives fédérées). Mais la mise en œuvre est importante. La même étude PMC a révélé que de nombreux systèmes n'exploitent pas pleinement la norme.
PostDicom propose un système PACS (Picture Archiving and Communication System) basé sur le cloud pour stocker, visualiser et partager des études d'imagerie et des documents cliniques. Quelques fonctionnalités clés de PostDICOM relatives aux métadonnées :
• Prise en charge des balises et des descriptions DICOM : Notre bibliothèque de ressources répertorie les « Modalités et balises DICOM », permettant aux utilisateurs d'accéder à des listes de balises et à des descriptions. Modalité et balises DICOM
• Intégration Api/Fhir : il prend en charge les interfaces API et FHIR (Fast Healthcare Interoperability Resources), permettant d'interroger les métadonnées par programmation, de les intégrer à d'autres systèmes et d'être analysées.
• Évolutivité du cloud et partage multisite : partage entre patients, médecins et institutions ; l'évolutivité illimitée signifie que les pipelines de mégadonnées deviennent réalisables.
• Traitement d'image avancé et prise en charge de la multimodalité : Bien que cela concerne les pixels, la prise en charge de modalités telles que la TEP-CT et la multisérie signifie que les métadonnées sont importantes (valeurs SUV, volumes de fusion, type de modalité) et disponibles pour l'analyse.
L'utilisation d'une plateforme telle que PostDICOM vous permet d'exploiter les métadonnées via des flux de travail structurés, des API et une architecture cloud.
Voici comment structurer le flux de travail, des archives brutes aux informations prêtes à être analysées.
 - Created by PostDICOM.jpg)
L'extraction et la normalisation constituent la première étape de l'exploitation des métadonnées DICOM à des fins d'analyse. Les bibliothèques telles que le package Python open source PyDICOM sont couramment utilisées pour analyser les fichiers DICOM et extraire les balises pertinentes, notamment les lignes et les colonnes d'images, les noyaux de convolution et les paramètres d'acquisition spécifiques à la modalité.
La gestion de l'hétérogénéité est cruciale, car différents fournisseurs utilisent souvent des balises privées ou des implémentations non standard. Une analyse analytique robuste, une logique de repli et des tables de mappage de balises complètes sont nécessaires pour garantir la cohérence entre les ensembles de données.
Une fois extraites, les métadonnées doivent être normalisées et mises en correspondance avec des ontologies et des structures standard, telles que les codes de modalité, les noms des fournisseurs, les catégories d'épaisseur des tranches et les formats de date et d'heure standardisés.
Enfin, les métadonnées structurées doivent être stockées dans un environnement de mégadonnées, tel qu'une base de données relationnelle, un stockage NoSQL ou un lac de données en colonnes, avec indexation pour permettre des requêtes rapides et efficaces.
Une fois extraites, les métadonnées doivent faire l'objet d'un contrôle qualité afin de garantir leur exactitude et leur fiabilité. Les champs manquants ou incohérents, tels que les valeurs d'épaisseur des tranches vides, les étiquettes de modalité incohérentes ou les UID d'instance d'étude dupliqués, doivent être identifiés et corrigés.
La confidentialité et l'anonymisation sont également essentielles à ce stade, car les métadonnées contiennent souvent des informations personnelles identifiables, notamment les noms, les identifiants et les dates des patients ; les outils et protocoles de désidentification sont essentiels.
La tenue de pistes d'audit complètes est une autre pratique importante, car elle permet de documenter la date à laquelle les métadonnées ont été extraites, les versions de l'analyseur qui ont été utilisées et les éventuelles erreurs rencontrées au cours du processus.
Les politiques de gouvernance devraient également définir des champs obligatoires et fournir des conseils sur la manière de gérer les ensembles de données existants ou incomplets afin de garantir l'exactitude et la conformité des analyses en aval.
L'étape suivante est l'indexation et l'ingénierie des fonctionnalités basées sur les métadonnées, qui transforment les métadonnées brutes en informations exploitables.
Cela implique la création d'indices et de filtres qui permettent aux chercheurs et aux analystes d'interroger des ensembles de données spécifiques, par exemple en récupérant tous les tomodensitogrammes thoraciques dont l'épaisseur de coupe est inférieure à 1,5 millimètre auprès d'un fournisseur particulier dans une plage de dates donnée.
L'ingénierie des fonctionnalités s'appuie sur cela en combinant des champs de métadonnées tels que le fournisseur, le modèle, la date d'acquisition, l'épaisseur de la tranche, le noyau de convolution, le protocole de contraste, la région corporelle, la dose de rayonnement et l'identifiant de l'institution dans des variables structurées adaptées à l'analyse.
Les métadonnées peuvent également être liées à des ensembles de données cliniques, reliant les données d'imagerie aux résultats, aux diagnostics ou aux traitements des patients. Ce couplage permet d'avoir une vision plus globale des données d'imagerie et de leur contexte clinique.
Une fois les métadonnées indexées et les fonctionnalités conçues, les analyses et la génération d'informations deviennent possibles.
Les analyses descriptives peuvent révéler les volumes d'études par modalité, fournisseur ou région, suivre les tendances des paramètres d'acquisition et mettre en évidence les erreurs ou les incohérences dans les pratiques d'imagerie. Les analyses comparatives permettent d'évaluer les protocoles d'acquisition entre les institutions, de détecter les écarts et d'identifier les analyses aberrantes qui peuvent nécessiter une attention particulière.
Pour les applications d'apprentissage automatique et d'IA, les métadonnées sont essentielles pour contrôler le changement de domaine, garantir que les ensembles de données d'entraînement et de test sont stratifiés de manière appropriée et combiner des fonctionnalités basées sur des pixels avec des variables de métadonnées structurées. Les tableaux de bord opérationnels peuvent ensuite exploiter ces données pour surveiller la charge de travail, évaluer les mesures d'assurance qualité et garantir la conformité des protocoles sur tous les sites.
Enfin, le feedback et l'amélioration continue complètent le cycle de vie des métadonnées. Les informations issues des analyses peuvent contribuer à affiner les protocoles d'acquisition et à normaliser les flux de travail afin d'améliorer la qualité globale des données.
Les nouvelles études d'imagerie et les nouvelles métadonnées doivent être ingérées en permanence, avec une surveillance des performances du magasin de métadonnées, des temps de requête et de l'intégrité des données. Les leçons apprises doivent être archivées pour capturer les champs de métadonnées prédictifs, corriger les lacunes ou les erreurs récurrentes et améliorer les pratiques de gouvernance.
Cette approche itérative garantit que les pipelines de métadonnées restent robustes, évolutifs et utiles pour les recherches futures, les applications d'IA et la prise de décisions opérationnelles.
• Variabilité entre le fournisseur et l'institution : étiquettes privées ou interprétations standard vagues.
• Métadonnées manquantes ou corrompues : les en-têtes des études plus anciennes peuvent être incomplets.
• Confidentialité et anonymisation des données : les données personnelles doivent être anonymisées pour les recherches multisites.
• Échelle et performances : des millions d'images nécessitent un traitement et un stockage efficaces.
• Changement de domaine et biais : les fournisseurs et protocoles dominants peuvent fausser les modèles d'IA.
• Problèmes de réglementation et de conformité : les déploiements multirégionaux peuvent impliquer la HIPAA, le RGPD ou des réglementations locales.
Les métadonnées DICOM constituent le squelette caché de l'analyse d'imagerie. Des plateformes telles que PostDICOM illustrent comment transformer une archive fragmentée de fichiers DICOM en un écosystème consultable, évolutif et piloté par des métadonnées. Si vous souhaitez découvrir PostDicom, nous vous encourageons à bénéficier de notre essai gratuit de 7 jours.